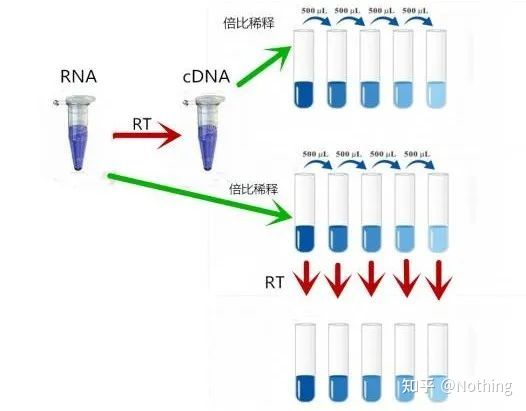
1. Compréhension initiale
A ce stade, nous avons besoin de comprendre certains concepts et termes, afin d'éviter de faire des erreurs devant nos seniors, telles que :
Q : Quelle est la différence entre la RT-PCR, la qPCR, la PCR en temps réel et la RT-PCR en temps réel ?
Réponse : La RT-PCR est une PCR à transcription inverse(PCR de transcription inverse, RT-PCR), qui est une variante largement utilisée de la réaction en chaîne par polymérase (PCR).Dans la RT-PCR, un brin d'ARN est rétrotranscrit en ADN complémentaire, qui est ensuite utilisé comme matrice pour l'amplification d'ADN par PCR.
PCR en temps réel et qPCR(Quantitative Rea-ltime-PCR) sont la même chose, les deux sont une PCR quantitative en temps réel, ce qui signifie que chaque cycle de PCR a des enregistrements de données en temps réel, de sorte que le nombre de modèles de départ peut être ajusté avec une analyse précise.
Bien que la PCR en temps réel (PCR quantitative fluorescente en temps réel) et la PCR de transcription inverse (PCR de transcription inverse) semblent être abrégées en RT-PCR, la convention internationale est la suivante : RT-PCR fait spécifiquement référence à la transcription inversePCR, la PCR en temps réel est généralement abrégée en qPCR (PCR quantitative en temps réel).
Et la RT-PCR en temps réel (RT-qPCR), c'est la transcription inverse PCR combinée à la technologie quantitative fluorescente: obtenez d'abord l'ADNc (RT) à partir de la transcription inverse de l'ARN, puis utilisez la PCR en temps réel pour l'analyse quantitative (qPCR).La plupart des laboratoires font de la RT-qPCR, c'est-à-dire de la recherche sur la régulation à la baisse de l'expression de l'ARN, donc la qPCR dont tout le monde parle au laboratoire se réfère en fait à la RT-qPCR, mais n'oubliez pas qu'il existe encore de nombreux tests ADN dans les applications cliniques.Analyse quantitative, telle que la détection du VHB par le virus de l'hépatite B.
Question : Après avoir lu beaucoup de PCR quantitative fluorescente, pourquoi le fragment amplifié devrait-il être contrôlé dans la plage de 80 à 300 pb ?
Répondre: La longueur de chaque séquence de gène est différente, certaines font plusieurs kb, d'autres des centaines de bp, mais il suffit d'exiger que la longueur du produit soit de 80 à 300 bp lors de la conception d'amorces, trop courtes ou trop longues ne conviennent pas à la détection par PCR quantitative fluorescente .Le fragment de produit est trop court pour être distingué de l'amorce-dimère.La longueur de l'amorce-dimère est d'environ 30 à 40 pb, et il est difficile de distinguer s'il s'agit d'une amorce-dimère ou d'un produit s'il est inférieur à 80 pb.Si le fragment de produit est trop long, dépassant 300 pb, il conduira facilement à une faible efficacité d'amplification et ne pourra pas détecter efficacement la quantité de gène.
Par exemple, lorsque vous comptez le nombre de personnes dans une salle de classe, vous n'avez qu'à compter le nombre de bouches.La même chose est vraie lorsque vous détectez des gènes, il vous suffit de détecter une certaine séquence d'un gène pour représenter la séquence entière fera l'affaire.Si vous voulez compter les gens, vous devez compter à la fois la bouche et le nez, les oreilles et les lunettes, et il est facile de faire des erreurs.
Pour élargir, dans la recherche biologique, il existe de nombreux cas de recherche d'un point à l'autre, car la séquence génétique de toute espèce est très longue, il est inutile et impossible de mesurer tous les fragments, comme le séquençage bactérien 16S, qui consiste à effectuer la séquence conservatrice de dosages de bactéries pour déduire le nombre d'une certaine population de bactéries.
Q : Quelle est la longueur optimale pour la conception d'amorces qPCR ?
Répondre: D'une manière générale, la longueur de l'amorce est d'environ 20-24bp, ce qui est mieux.Bien sûr, nous devons faire attention à la valeur TM de l'amorce lors de la conception de l'amorce, car celle-ci est liée à la température de recuit optimale.Après de nombreuses expériences, il a été prouvé que 60°C est une meilleure valeur TM.Si la température de recuit est trop basse, cela conduira facilement à une amplification non spécifique.Si la température de recuit est trop élevée, l'efficacité d'amplification sera relativement faible, le pic de la courbe d'amplification commencera plus tard et la valeur CT sera retardée.
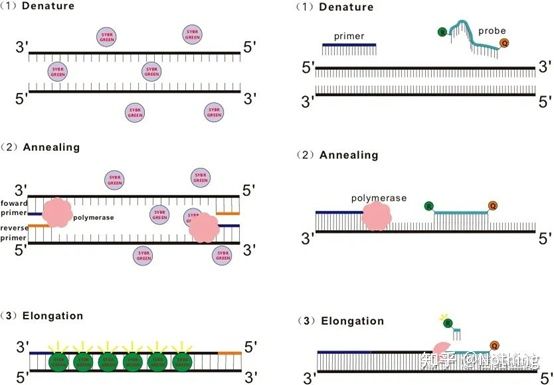
Q : En quoi la méthode du colorant diffère-t-elle de la méthode de la sonde ?
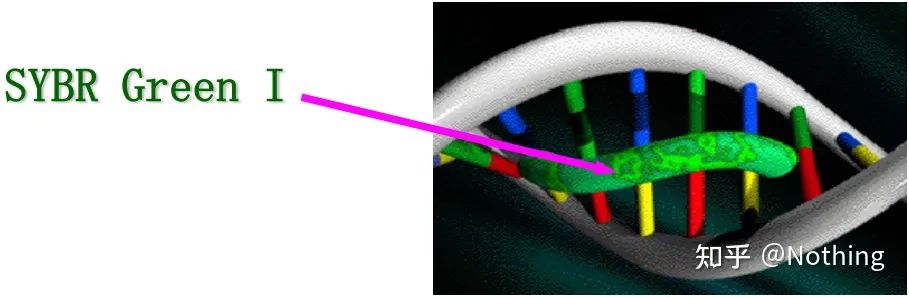
Réponse : Méthode de teintureCertains colorants fluorescents, tels que SYBR Green Ⅰ, PicoGreen, BEBO, etc., n'émettent pas de lumière par eux-mêmes, mais émettront une fluorescence après s'être liés au sillon mineur de l'ADN double brin.Par conséquent, au début de la réaction PCR, la machine ne peut pas détecter le signal fluorescent.Lorsque la réaction atteint l'étape d'hybridation-extension, le double brin est ouvert, et un nouveau brin est synthétisé sous l'action de l'ADN polymérase, et la molécule fluorescente se lie au petit sillon d'ADNdb.Au fur et à mesure que le nombre de cycles de PCR augmente, de plus en plus de colorants sont combinés avec de l'ADN double brin, et le signal fluorescent est également continuellement amélioré.La méthode de la teinture est principalement utilisée dans la recherche scientifique.
PS : Soyez prudent lorsque vous faites l'expérience, le colorant doit être combiné avec de l'ADN humain, faites attention à le transformer en une personne fluorescente.
Méthode colorant (gauche) Méthode sonde (droite)
PS : Soyez prudent lorsque vous faites l'expérience, le colorant doit être combiné avec de l'ADN humain, faites attention à le transformer en une personne fluorescente.
SYBR Green Ⅰ se lie au petit sillon de l'ADN
Méthode de sondeLa sonde Taqman est la sonde d'hydrolyse la plus couramment utilisée.Il existe un groupe fluorescent à l'extrémité 5 'de la sonde, généralement FAM, et la sonde elle-même est une séquence complémentaire du gène cible.Il y a un groupe d'extinction fluorescent à l'extrémité 3 '.Selon le principe du transfert d'énergie de résonance de fluorescence (Förster resonance energy transfer, FRET), lorsque le groupe fluorescent rapporteur (molécule fluorescente donneuse) et le groupe fluorescent quenching (molécule fluorescente acceptrice) sont excités Lorsque les spectres se chevauchent et que la distance est très proche (7-10nm), l'excitation de la molécule donneuse peut induire la fluorescence de la molécule acceptrice, tandis que l'autofluorescence est affaiblie.Par conséquent, au début de la réaction PCR, lorsque la sonde est libre et intacte dans le système, le groupe fluorescent rapporteur n'émettra pas de fluorescence.Lors de l'annelage, l'amorce et la sonde se lient à la matrice.Au cours de l'étape d'extension, la polymérase synthétise en continu de nouvelles chaînes.L'ADN polymérase a une activité d'exonucléase 5'-3'.Lorsqu'elle atteint la sonde, l'ADN polymérase hydrolyse la sonde de la matrice, sépare le groupe fluorescent rapporteur du groupe fluorescent extincteur et libère le signal fluorescent.Puisqu'il existe une relation biunivoque entre la sonde et la matrice, la méthode de la sonde est supérieure à la méthode du colorant en termes de précision et de sensibilité du test.La méthode de la sonde est principalement utilisée dans le diagnostic.
Q : Qu'est-ce que la quantification absolue ?Qu'est-ce que la quantification relative ?
Répondre: La quantification absolue fait référence au calcul du nombre de copies initiales de l'échantillon à tester par qPCR, comme le nombre de virus VHB dans 1 ml de sang.Le résultat obtenu par quantification relative est le changement de la quantité du gène cible dans un échantillon spécifique par rapport à un autre échantillon de référence, et l'expression du gène est régulée à la hausse ou à la baisse.
Q : La quantité d'extraction d'ARN, l'efficacité de la transcription inverse et l'efficacité de l'amplification affecteront-elles les résultats expérimentaux ?
Q : Le stockage des échantillons, les réactifs d'extraction, les réactifs de transcription inverse et les consommables transmettant la lumière affecteront-ils les résultats expérimentaux ?
Q : Quelle méthode peut corriger les données expérimentales ?
Concernant ces problèmes, nous les décrirons en détail dans les sections avancées et avancées ci-dessous.
2. Connaissances avancées
En ce qui concerne la PCR quantitative fluorescente en temps réel, nous devons reconnaître la réalité que des milliers d'articles de recherche scientifique sont publiés chaque année, parmi lesquels la technologie PCR quantitative fluorescente n'est pas un petit nombre.
S'il n'y a pas de norme commune pour mesurer l'expérience de PCR quantitative fluorescente, les résultats peuvent varier considérablement.Pour le même gène de la même espèce, avec la même méthode de traitement, les résultats de détection varieront également considérablement, et il sera difficile pour les retardataires de répéter les mêmes résultats.Toi Personne ne sait qui a raison et qui a tort.
Cela signifie-t-il que la PCR quantitative fluorescente est une technologie de triche ou une technologie peu fiable ?Non, c'est parce que la PCR quantitative fluorescente est plus sensible et plus précise, et une petite opération erronée produira des résultats complètement opposés.Une petite perte est à des milliers de kilomètres.L'auteur de l'article peut être torturé à plusieurs reprises par les critiques.Dans le même temps, les examinateurs de la revue sont également difficiles à choisir parmi différents résultats expérimentaux.
Dans l'ensemble, ce qui indique un manque de consensus dans les expériences de PCR en temps réel.À cette fin, des scientifiques chevronnés de l'industrie ont commencé à formuler des normes,obligeant les contributeurs à fournir certains détails expérimentaux et de traitement de données nécessaires (y compris les données nécessaires) dans l'article pour répondre à ces normes .
Les examinateurs peuvent juger de la qualité de l'expérience en lisant ces détails ;les futurs lecteurs peuvent également l'utiliser pour répéter l'expérience ou améliorer l'expérience.Ensuite, les résultats expérimentaux ainsi obtenus sont riches en informations, de grande qualité et utilisables.
MIBBI (Informations minimales pour les enquêtes biologiques et biomédicales -http://www.mibbi.org) il s'est avéré.MIBBI est un projet qui fournit des normes pour les expériences.Il est publié dans la nature.Ce projet vise diverses expériences biologiques, y compris la biologie cellulaire, Microarray, qPCR dont nous allons discuter maintenant, etc., et prévoit chaque type d'expérience lors de la soumission de manuscrits.Ces informations doivent être fournies à tout moment.
Dans le projet MIBBI, il y a deux articles liés à la PCR quantitative fluorescente, à savoir:
·RDML (Real-Time PCR Data Markup Language) - un langage structuré et un guide de rapport pour les données PCR quantitatives en temps réel ;
·MIQE (Informations minimales pour la publication d'expériences PCR quantitatives en temps réel) - informations minimales pour la publication d'articles sur les expériences PCR quantitatives en temps réel.
Parlons d'abord de RDML, la spécification terminologique.
S'il n'y a pas de définition standard pour tout, il est impossible de poursuivre la discussion, c'est pourquoi l'explication des termes est si importante dans l'examen.
La terminologie utilisée dans l'expérience de PCR quantitative fluorescente comprend le contenu suivant.QIAGEN a fait le meilleur résumé pour nous.Les suivants sont tous secsmarchandises .
Courbe d'amplification
La courbe d'amplification fait référence à la courbe réalisée au cours du processus de PCR, avec le numéro de cycle en abscisse et l'intensité de fluorescence en temps réel pendant la réaction en ordonnée.
Une excellente courbe d'amplification doit avoir les caractéristiques suivantes: la ligne de base est plate ou légèrement diminuée, et il n'y a pas de tendance évidente à la hausse ;le point d'inflexion de la courbe est clair, et la pente de la phase exponentielle est proportionnelle au rendement d'amplification.Plus la pente est grande, plus le rendement d'amplification est élevé ;la courbe d'amplification globale Le parallélisme est bon, indiquant que l'efficacité d'amplification de chaque tube est similaire ;la phase exponentielle de la courbe d'amplification des échantillons à faible concentration est évidente.
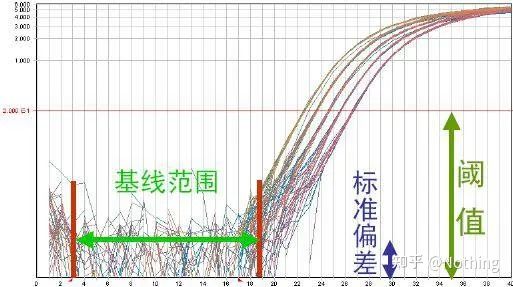
Base de référence (Base de référence)
La ligne de base est le niveau de bruit du début du cycle, généralement mesuré entre le 3e et le 15e cycle, car l'augmentation de la valeur de fluorescence provoquée par le produit d'amplification ne peut pas être détectée pendant cette période.Le nombre de cycles utilisés pour calculer la ligne de base peut varier et peut devoir être réduit si des quantités élevées de matrice sont utilisées ou si le niveau d'expression du gène cible est élevé.
Le réglage de la ligne de base nécessite l'affichage des données de fluorescence à partir de la courbe d'amplification de linéarité.La ligne de base est définie de manière à ce que la croissance de la courbe d'amplification commence par un nombre de cycles supérieur au nombre maximal de cycles de base.Les lignes de base doivent être définies individuellement pour chaque séquence cible.Les valeurs moyennes de fluorescence détectées dans les premiers cycles doivent être soustraites des valeurs de fluorescence obtenues dans les produits amplifiés.Les dernières versions de divers logiciels de PCR en temps réel permettent une optimisation automatique des paramètres de référence pour les échantillons individuels.
Au cours des premiers cycles de la réaction d'amplification PCR, le signal de fluorescence ne change pas beaucoup.L'approche d'une ligne droite s'appelle la ligne de base, mais si nous regardons attentivement les premiers cycles, nous voyons que dans la ligne de base se trouve ce qui se passe dans l'image ci-dessous.
Arrière-plan L'arrière-plan fait référence à
la valeur de fluorescence non spécifique dans la réaction .Par exemple : extinction de fluorescence inefficace ;ou un grand nombre de matrices d'ADN double brin en raison de l'utilisation de SYBR Green.Les composants de fond du signal sont mathématiquement supprimés par l'algorithme du logiciel PCR en temps réel.
Signal de journaliste
Le signal rapporteur fait référence au signal fluorescent généré par SYBR Green ou des sondes spécifiques à la séquence marquées par fluorescence pendant la PCR en temps réel.
Signal de rapporteur normalisé (RN)
RN fait référence à l'intensité de fluorescence du colorant rapporteur divisée par l'intensité de fluorescence du colorant de référence passif mesurée à chaque cycle.
Colorant de référence passif
Dans certains PCR en temps réel,le colorant fluorescent ROX est utilisé comme référence interne pour normaliser le signal fluorescent.Il corrige les variations dues à un pipetage imprécis, à la position des puits et aux fluctuations de fluorescence puits par puits.
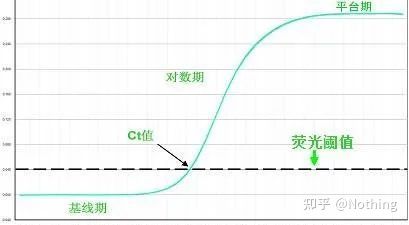
Le seuil de fluorescence (seuil)
a été ajustée au-dessus de la valeur de fond et significativement en dessous de la valeur plateau de la courbe d'amplification.Il doit se situer dans la région linéaire de la courbe d'amplification, représentant la plage log-linéaire de détection PCR.Les seuils doivent être définis dans la vue de la courbe d'amplification log afin que la phase log-linéaire de la PCR soit facilement identifiable.S'il existe plusieurs gènes cibles dans la PCR en temps réel, le seuil doit être défini pour chaque cible .Généralement, le signal de fluorescence des 15 premiers cycles de réaction PCR est utilisé comme signal de fond de fluorescence, et le seuil de fluorescence est 10 fois l'écart type du signal de fluorescence des 3 à 15 premiers cycles de PCR, et le seuil de fluorescence est défini dans la phase exponentielle de l'amplification PCR.En général, chaque instrument a son seuil de fluorescence défini avant utilisation.
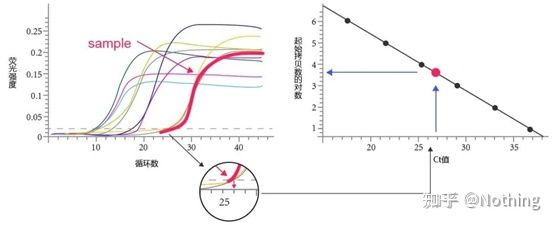
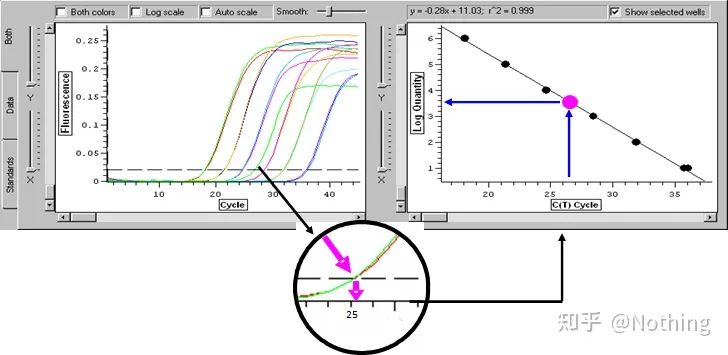
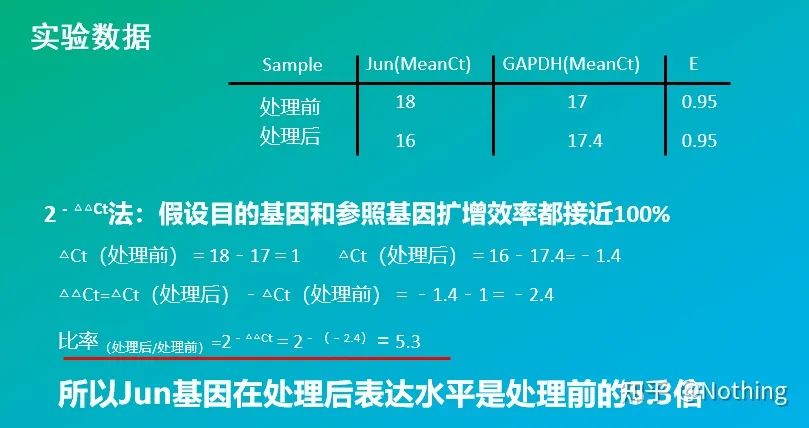
Cycle Threshold (CT) ou Crossing Point (CP)
Le cycle auquel la courbe d'amplification franchit le seuil (c'est-à-dire le point auquel la détection de fluorescence augmente de manière significative).CT peut être une fraction et la quantité de modèle de départ peut être calculée.La valeur CT représente le nombre de cycles subis lorsque le signal fluorescent dans chaque tube de réaction PCR atteint le seuil défini.Il existe une relation linéaire entre la valeur CT de chaque modèle et le logarithme du nombre de copies initiales du modèle, leplus le nombre de copies initiales est élevé, plus la valeur CT est petite et vice versa.Une courbe standard peut être réalisée en utilisant un standard avec un nombre de copies initial connu, dans laquelle l'abscisse représente la valeur CT et l'ordonnée représente le logarithme du nombre de copies initial.Par conséquent, tant que la valeur CT de l'échantillon inconnu est obtenue, le nombre de copies initial de l'échantillon peut être calculé à partir de la courbe standard.
Valeur ΔCT
La valeur ΔCT décritla différence entre le gène cible et la valeur CT du gène endogène de référence correspondant, tel qu'un gène de ménage, et est utilisé pour normaliser la quantité de modèle utilisée :
⇒ΔCT = CT (gène cible) – CT (gène de référence endogène)
Valeur ΔΔCT
La valeur ΔΔCT décrit la différence entre la valeur moyenne ΔΔCT d'un échantillon d'intérêt (par exemple, des cellules stimulées) et la valeur moyenne ΔΔCT d'un échantillon de référence (par exemple, des cellules non stimulées).L'échantillon de référence est également appelé échantillon d'étalonnage et tous les autres échantillons sont normalisés à celui-ci pour une quantification relative :
⇒ΔΔCT = ΔCT moyen (échantillon d'intérêt) – ΔCT moyen (échantillon de référence)
Gènes de référence endogènes (gènes de référence endogènes)
Les niveaux d'expression des gènes de référence endogènes, tels que les gènes de ménage (housekeeping genes), ne diffèrent pas entre les échantillons.La comparaison des valeurs CT du gène de référence au gène cible permet de normaliser le niveau d'expression du gène cible à la quantité d'ARN ou d'ADNc d'entrée (voir la section sur les valeurs ΔCT ci-dessus).
Gènes de référence internes corrects pourla dégradation possible de l'ARN ou la présence d'inhibiteurs d'enzymes dans les échantillons d'ARN, ainsi que les variations de la teneur en ARN, l'efficacité de la transcription inverse, la récupération des acides nucléiques et la manipulation des échantillons.Pour sélectionner le ou les gènes de référence optimaux, nous avons modifié l'algorithme pour permettre sa sélection de la référence optimale en fonction du cadre expérimental.
Contrôle interne
Une séquence de contrôle qui est amplifiée dans la même réaction que la séquence cible et sondée avec une sonde différente (c'est-à-dire, en réalisant une PCR duplex).Des contrôles internes sont souvent utilisés pour exclure les amplifications ayant échoué, par exemple lorsque la séquence cible n'est pas détectée.
Échantillon d'étalonnage
Échantillon de référence (par exemple, ARN purifié d'une lignée cellulaire ou d'un tissu) utilisé dans la quantification relative pour comparer tous les autres échantillons afin de déterminer le niveau d'expression relatif d'un gène.L'échantillon d'étalonnage peut être n'importe quel échantillon, mais il s'agit généralement d'un témoin (par exemple, un échantillon non traité ou un échantillon du temps zéro de l'expérience).
Témoins positifs
utiliser des réactions de contrôle avecquantités connues de modèle.Les contrôles positifs sont souvent utilisés pour vérifier qu'un jeu d'amorces ou un jeu d'amorces-sondes fonctionne correctement et que la réaction est correctement configurée.
Aucun contrôle de modèle (NTC)
Une réaction de contrôle qui contient tous les composants nécessaires de la réaction d'amplification à l'exception de la matrice, qui est généralement remplacée par de l'eau.L'utilisation de NTC peut trouver la contamination causée par une contamination par un réactif ou un ADN étranger, garantissant ainsi l'authenticité et la fiabilité des données de détection.L'amplification du contrôle NTC indique une contamination.
Pas de contrôle RT (NRT)
Le processus d'extraction d'ARN peut contenir de l'ADN génomique résiduel, qui est extrêmement nocif et est le coupable affectant la qualité des données et l'ennemi naturel de la qPCR, donc lors de la conception d'expériences, il doit être conçu pour amplifier uniquement la détection d'ARN.Il existe deux façons, l'une consiste à concevoir des amorces à travers les introns, l'autre consiste à éliminer complètement l'ADN, laquelle est la meilleure, ce qui sera discuté plus tard.Le contrôle NTR est un miroir magique pour détecter la pollution de l'ADN.S'il y a amplification, c'est qu'il y a pollution.
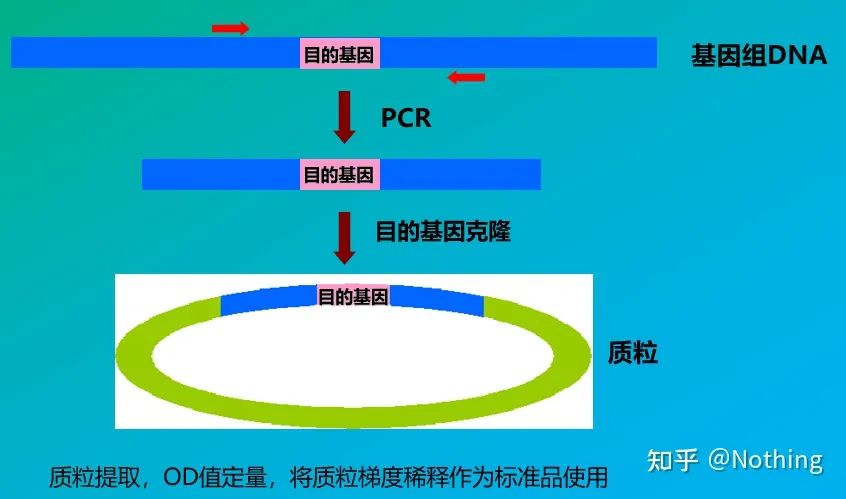
Normes
Les standards sont des échantillons de concentration ou de nombre de copies connus qui sont utilisés pour construire une courbe standard.Afin d'assurer la stabilité du standard, le fragment de gène est généralement cloné dans le plasmide et utilisé comme standard.
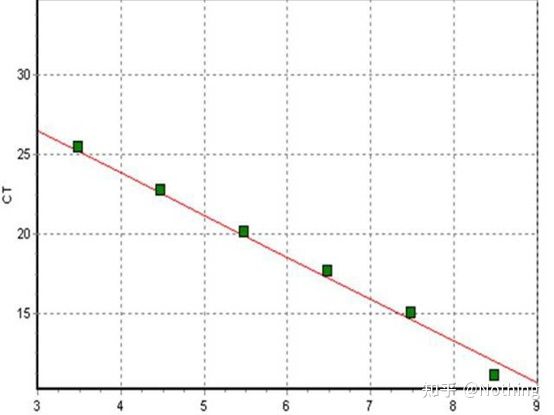
La courbe standard
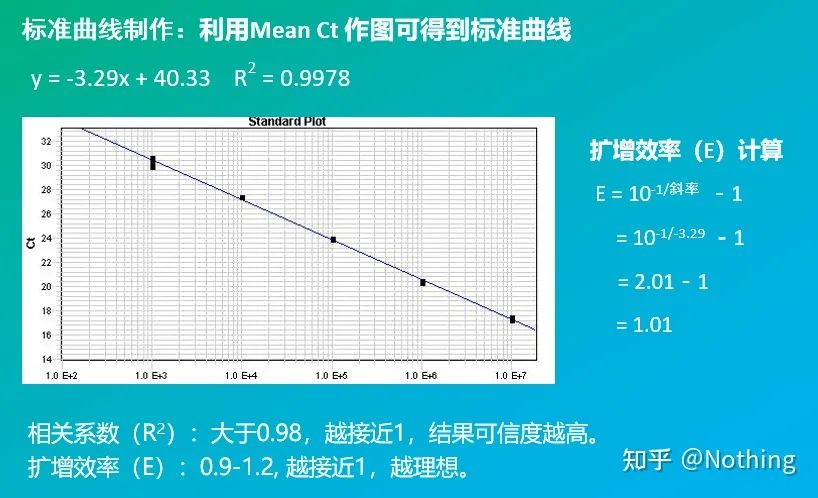
est généralement dilué dans au moins 5 gradients de concentration avec le produit standard selon le rapport de doublement, et 5 points sont dessinés dans les coordonnées de la valeur CT et du nombre de copies, et les points sont connectés pour former une ligne pour générer une courbe standard.Pour chaque courbe standard, sa validité doit être vérifiée.La valeur de la pente se situe entre –3,3 et –3,8, et chaque concentration est effectuée en triple.Les points qui sont significativement différents des autres points doivent être éliminés.La valeur CT de l'échantillon à tester est introduite dans la courbe standard et le niveau d'expression de l'échantillon à tester peut être calculé.
La valeur CT de l'échantillon à tester est introduite dans la courbe standard et le nombre de copies initiales de l'échantillon à tester peut être calculé.
Efficacité et pente
La pente de la courbe standard représente l'efficacité de la PCR en temps réel.
·Une pente de -3,322 indique que l'efficacité de l'amplification PCR est de 1, soit 100 % d'efficacité, et que la quantité de produit PCR double à chaque cycle.
·Une pente inférieure à -3,322 (par exemple, -3,8) indique une efficacité PCR
·Une pente supérieure à -3,322 (par exemple, -3,0) indique que l'efficacité de la PCR semble être supérieure à 100 %, ce qui est curieux, comment un cycle de PCR pourrait-il générer plus du double du produit amplifié ?Cette situation se produit dans la phase non linéaire de la réaction PCR, c'est-à-dire qu'il y a une grande quantité d'amplification non spécifique.
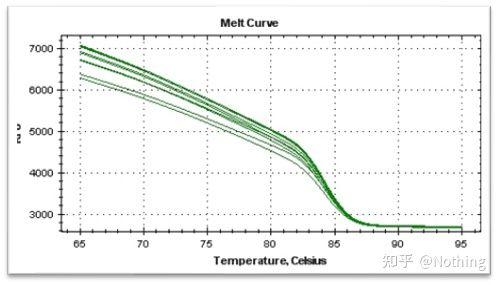
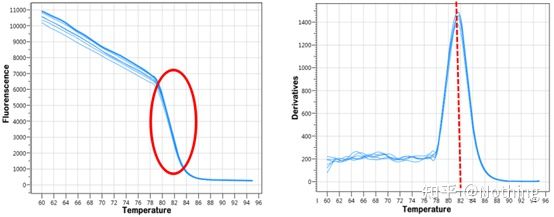
courbe de fusion
Une fois l'amplification qPCR terminée, le produit PCR est chauffé.Lorsque la température augmente, le produit d'amplification double brin fond progressivement, entraînant une diminution de l'intensité de fluorescence.Lorsqu'une certaine température (Tm) est atteinte, un grand nombre de produits vont fondre.La fluorescence chute fortement.Différents produits de PCR ont différentes valeurs de Tm et différentes températures de fusion, de sorte que la spécificité de la PCR peut être identifiée.
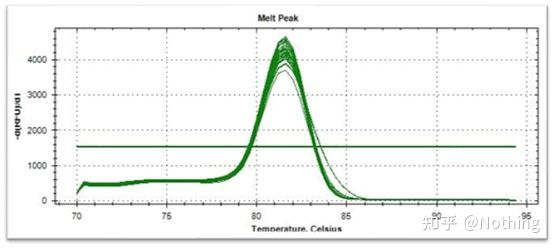
Courbe de fusion (courbe dérivée)
La courbe de fusion est dérivée pour former une carte de pic, qui peut afficher plus intuitivement la situation des fragments de produit PCR.Étant donné que la température de fusion est la valeur Tm du fragment d'ADN, certains paramètres qui affectent la valeur Tm du fragment d'ADN peuvent être jugés, tels que la taille du fragment, la teneur en GC, etc. D'une manière générale, selon nos principes de conception d'amorce,la longueur du produit amplifié est comprise entre 80 et 300 pb, la température de fusion doit donc être comprise entre 80°C et 90°C.
Interprétation de la courbe de fusion: Si le seul pic principal apparaît entre 80°C-90°C, cela signifie que la PCR quantitative fluorescente est parfaite ;si le pic principal apparaît entre 80°C et 90°C et que divers pics apparaissent en dessous de 80°C, le dimère d'amorce est fondamentalement considéré.Vous pouvez essayer d'augmenter la température de recuit pour le résoudre ;si le pic principal apparaît entre 80 °C et 90 °C et que le pic divers réapparaît lorsque la température augmente, on considère fondamentalement qu'il y a contamination par l'ADN et que l'ADN doit être retiré au stade initial de l'expérience.
Bien sûr, il reste encore quelques situations anormales, qui seront décomposées une à une ci-dessous.
3. Connaissances avancées
Pour faire qPCR, je dois dire MIQE,Informations minimalespour la publication deQuantitatifPcr en temps réelExpériences - les informations minimales pour publier des articles sur la PCR quantitative en temps réelexpériences .Afin de simplifier la compréhension de chacun, nous allons simplifier le contenu clé.
Vous pouvez rechercher le texte original du MIQE sur Internet, et le plus important est qu'il stipule leliste de contrôle des données qui doit être fournie lors de la publication d'un article .
Les examinateurs peuvent juger de la qualité de l'expérience en lisant ces détails ;les futurs lecteurs peuvent également l'utiliser pour répéter ou améliorer l'expérience.
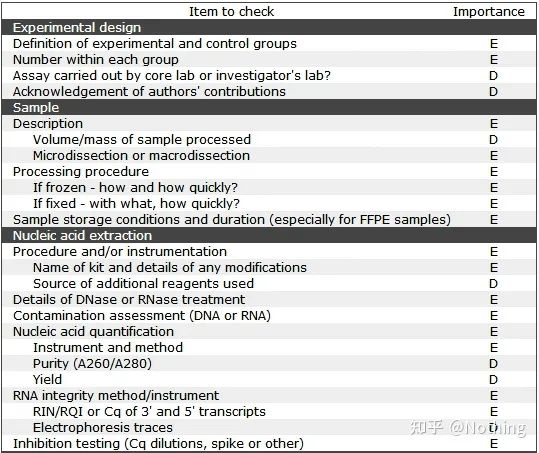
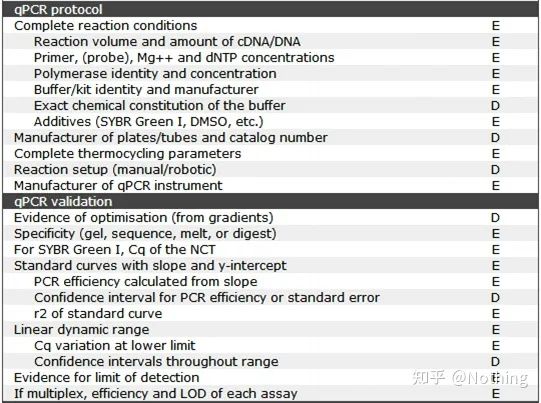
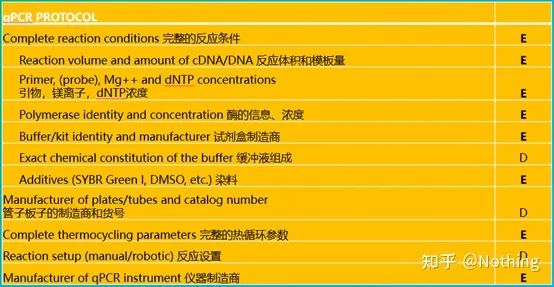
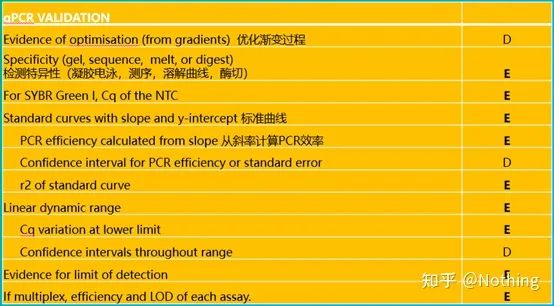
Il convient de noter que dans cette liste, l'importance de chaque liste est marquée respectivement par E ou D.Qu'est-ce que ça veut dire?E : informations essentielles (doivent être soumises) ;D : informations souhaitables (fournir autant que possible).
MIQE (1)—Design expérimental
De nombreux scumbags qui ont terminé leur défense après avoir terminé leurs études supérieures ne sauront pas comment concevoir une expérience de manière autonome, ouvrir leurs cahiers et faire ce que l'enseignant leur dit de faire.En conséquence, la conception expérimentale n'était pas rigoureuse et la rédaction du magazine a déclaré qu'elle voulait inventer cette image et cette image, alors elle l'a fait dans un état second.C'est comme ça que sont faits les scumbags !
Plus près de nous, le premier principe de l'expérience est de déterminerla rigueur de la logique expérimentale.La chose la plus fondamentale est la conception expérimentale, et la chose la plus importante à propos de la conception expérimentale est de savoir comment définir l'échantillon cible, l'échantillon de référence (contrôle) et le nombre de répétitions, de sorte que les données expérimentales puissent être référencées, comparables et convaincantes.
L'échantillon ciblefait référence à l'échantillon qui nous oblige à détecter le gène cible après un certain traitement.L'échantillon de référenceest l'échantillon sans aucun traitement, qui est souvent appelé type sauvage en biologie.
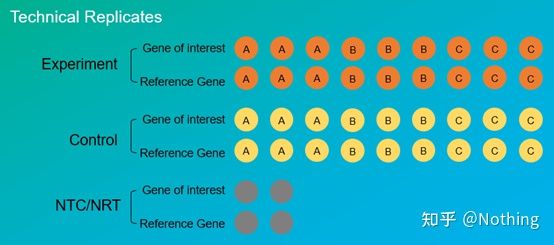
Répétitions expérimentalesSont très importants.Généralement, le nombre de répliques convaincantes doit être supérieur à trois.Il faut distinguer ce qui est la réplication biologique et ce qui est la réplication technique.
Répliques biologiques: La même expérience de vérification faite avec des matériaux différents (temps, plantes, lots, plaques de réaction).
Duplication biologique
Prenons l'exemple du traitement pesticide du poivre.Nous voulons pulvériser des pesticides sur les trois plantes d'ABC, puis les trois plantes d'ABC sont trois répétitions biologiques, et ce sont la même expérience de vérification réalisée avec des matériaux différents.Mais en tant qu'expérience, un contrôle est certainement nécessaire, nous pouvons donc pulvériser l'une des branches de la plante A pour former un groupe expérimental de plante A, et ne pas pulvériser les autres branches de la plante A pour former un groupe de contrôle.Faites de même pour B et C.
Répliques techniques (répliques techniques): Il s'agit d'une expérience répétée conçue pour éviter les erreurs causées par le fonctionnement, qui est en fait un trou en double inclus dans le même matériau.Les traitements et les contrôles doivent avoir des paramètres répliqués (minimum trois) du gène cible et du gène de référence interne.
Répétition technique
Reprenons l'exemple du poivre traité avec des pesticides.Pour le groupe expérimental de la plante A, nous avons fait trois trous PCR de 1, 2 et 3 pour son gène cible et son gène de référence interne respectivement, de manière à prendre la moyenne après la détection.Pour le contrôle de la plante A, les groupes sont également traités de la même manière.De même, faites le même traitement pour les plantes B et C.C'est de la répétition technique.
Il est à noter quece qui entre dans les statistiques, c'est la répétition biologique, et la répétition technique, c'est tester s'il y a des phénomènes aléatoires dans le processus expérimental, de manière à rendre crédibles les résultats expérimentaux, c'est-à-dire à éviter les erreurs en prenant leur moyenne comme on dit souvent.
Contrôles négatifs—NTC et NRT
NTC (contrôle sans modèle), un contrôle sans matrice, est utilisé pour vérifier si le matériel expérimental est contaminé.Généralement, l'eau est utilisée comme modèle.S'il y a une réaction fluorescente, cela indique qu'une contamination par l'acide nucléique s'est produite dans le laboratoire.
Ces pollutions proviennent de : eau impure, réactifs non qualifiés contenant de l'ADN endogène, pollution des amorces, pollution des équipements de laboratoire, pollution des aérosols, etc., nécessité d'utiliser des piégeurs de RNase et des inhibiteurs de RNase.La pollution par les aérosols est la plus difficile à trouver.Imaginez que votre laboratoire ressemble à du smog, avec divers acides nucléiques en suspension dans l'air.
NRT (Non-Reverse Transcriptase), le contrôle sans transcription inverse, est l'ARN transcrit non inverse en tant que contrôle négatif, qui est le contrôle du résidu d'ADNg.
Lors de l'expression génique, la quantité d'ARN est détectée en détectant la quantité d'ADNc après la transcription inverse.S'il y a un résidu d'ADNg lorsque l'ARN est purifié, cela entraînera des erreurs dans les résultats expérimentaux, car les résultats réels obtenus sont l'ADNg et l'ADNc.Au niveau agrégé, pas seulement l'ADNc, l'ADNg doit être complètement éliminé lors de l'extraction de l'ARN.
MIQE (2)—exemple d'informations
Les soi-disant informations sur l'échantillon signifient que lorsque nous publions un article sur la qPCR, nous devons expliquer clairement les informations sur l'échantillon, ce qui est une partie indispensable de l'article.De même, lorsque nous traitons des échantillons, nous devons également réglementer nos propres opérations pour garantir la validité des échantillons.
La description de l'échantillon n'est qu'un résultat, et nous devrions prêter plus d'attention aux matériaux prélevés pendant toute l'expérience.
Sélection de matériaux expérimentaux
Échantillons de sang - choisissez du sang frais, pas plus de 4 heures.Échantillons de cellules - choisissez de collecter des cellules fraîches dans une période de croissance vigoureuse.Tissu animal—Choisissez des tissus frais à croissance vigoureuse.Tissu végétal - Choisissez des tissus frais et jeunes.
Vous avez dû remarquer qu'il y a un mot clé dans ces quelques phrases : frais.
Pour les échantillons ci-dessus, le kit le meilleur, le plus rentable et le plus stable du marché est le kit de Foregene, qui peut extraire rapidement et facilement leur ADN et leur ARN.
Kit d'isolement d'ARN total cellulaire
Kit d'isolement d'ARN total animal
Kit d'isolement d'ARN total de plantes
Kit d'isolement d'ARN total de plantes Plus
Stockage de matériel expérimental
D'une manière générale, nous ne recommandons pas de conserver les échantillons, si les conditions le permettent.Cependant, de nombreux amis ne peuvent pas mener d'expériences immédiatement après l'échantillonnage, et certains doivent même transporter des réservoirs d'azote liquide sur le terrain pour l'échantillonnage.
Pour ce genre d'ami qui travaille dur, je peux seulement dire que vous ne comprenez pas les consommables réactifs.Aujourd'hui, de nombreuses sociétés de réactifs consommables produisent des réactifs qui peuvent stocker des échantillons d'ARN à température ambiante, et vous pouvez choisir de les utiliser.La méthode de stockage conventionnelle est le stockage d'azote liquide, à l'aide d'un petit réservoir d'azote liquide facile à transporter.Après avoir ramené l'échantillon au laboratoire, le conserver dans un réfrigérateur à -80°C.
Pour les expériences impliquant de l'ARN, le principe des six mots doit être suivi :basse température, pas d'enzymes,etrapide .
Le concept de basse température est facile à comprendre ;sans enzymes, la RNase est partout dans le monde dans lequel nous vivons (sinon vous auriez été tué par le VIH), alors comment éviter la RNase lorsque vous faites des expériences est un concept très important ;rapide,Il n'y a pas de Kung Fu dans le monde qui ne puisse être cassé, seule la vitesse ne peut pas être cassée.
Par conséquent, dans un sens, plus le temps d'extraction est court, meilleur est le kit.PourquoiPrégèneLe kit met l'accent sur la vitesse, car ils la connaissent bien.
PS : Certaines filles font des expériences très minutieuses, mais elles ne sont pas aussi bonnes qu'un slam dunk après plusieurs années de travail.Ils pensent que Dieu est injuste, se plaint des autres et cherche la vie.En fait, elle ne l'a pas compris.Il n'a pas bien protégé l'ARN et le joueur de slam dunk était agile.Quand il faisait l'expérience, il pensait qu'il finirait le slam dunk avec trois fois, cinq fois et deux divisions, mais il a bien fait l'expérience.
Note: Plus lent, plus de chances d'invasion de RNase.Comment s'entraîner pour être rapide ?Il n'y a aucun moyen, il suffit de pratiquer plus.
Pour différentes expériences et différents échantillons, il est encore nécessaire de lire plus de littérature et de choisir une méthode appropriée pour le traitement.Pour le processus de collecte et de stockage des échantillons, MIQE exige qu'il soit clairement écrit dans l'article, afin que les examinateurs puissent examiner la fiabilité de l'article, et il est également pratique pour les jeunes étourdis de répéter votre expérience.
Bien que les expériences biologiques soient difficiles, elles sont haut de gamme.Si vous ne faites pas attention, vous pouvez renverser le monde.Par exemple, faire du SRAS une crise biochimique ou fabriquer du riz hybride pour sauver 1,3 milliard de personnes.L'image ci-dessous est une expérience chimique, vous devriez comprendre à quel point vous êtes fier de votre recherche rien qu'en regardant son apparence de bite.Oubliez ça, ne le noircissez pas.
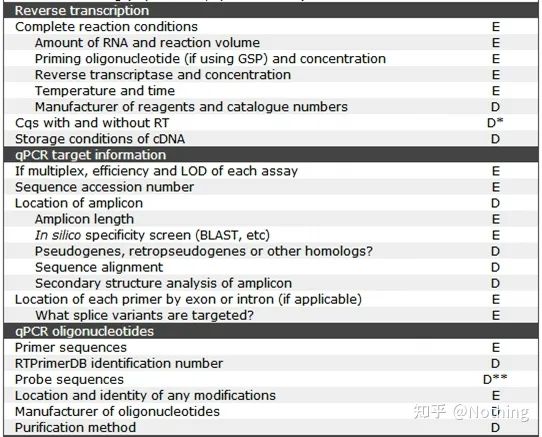
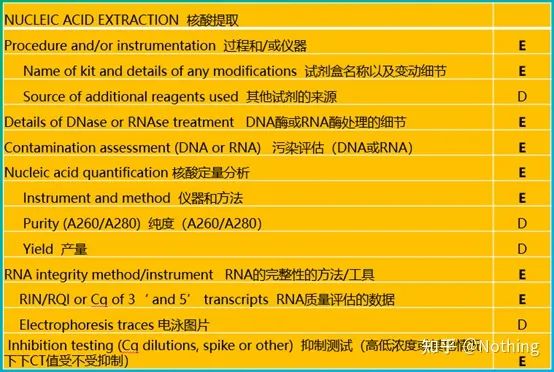
MIQE (3) – extraction d'acide nucléique.
L'extraction d'acide nucléique est un grand événement, et toutes les expériences de biologie moléculaire commencent par l'extraction d'acide nucléique.Tout d'abord, copions le contenu de MIQE sur l'extraction des acides nucléiques.
En regardant ce formulaire, vous ne pouvez pas rester à la surface.La forme est un dogme.Pour être un bon étudiant, vous devez vous demander pourquoi.Le contenu essentiel de ce tableau est : Poursuivrela pureté, l'intégrité, la cohérence et la quantité d'extraction d'ARN .
La première partie duprocédé ou instrument est l'étape d'extraction de l'acide nucléique.Si vous utilisez un extracteur automatique d'acide nucléique pour extraire (avancé, veuillez me contacter pour l'achat), vous devez indiquer le nom du modèle de l'instrument.
Le nom du kit et
quel kit a été utilisé pour les détails du changement, quels réactifs spéciaux ont été ajoutés ou quelles opérations spéciales ont été effectuées doivent être expliqués clairement afin que d'autres puissent facilement répéter votre expérience.
Certaines personnes ajoutent des réactifs spéciaux lors de l'extraction d'échantillons spéciaux, pensant que c'est leur arme secrète et ne le disent pas aux autres.Tout en gardant le secret, ils perdent également la possibilité de faire briller votre article.Ne sois pas intelligent, tu dois être plus honnête que le vieux Zhang dans la recherche scientifique, si tu veux être intelligent, l'article te rendra stupide.
doit se souvenir du numéro de produit du kitlorsque vous commandez le kit et écrivez l'article.Il y a généralement deux numéros sur le kit : Cat—numéro de catalogue (numéro de produit, numéro d'article), Lot—numéro de lot du produit (utilisé pour indiquer de quel lot provient le produit).
De plus, le numéro CAS est souvent utilisé lors de la commande de réactifs biochimiques, et je vais le vulgariser ensemble.Le numéro CAS est le numéro attribué par l'American Chemical Society à chaque nouveau médicament chimique.Généralement, trois nombres sont reliés par un tiret.Numéro CAS de Rushui : 7732-18-5.Les produits chimiques ont souvent plusieurs alias, mais le numéro CAS est unique.Lorsque vous commandez un médicament, vous pouvez d'abord vérifier son numéro CAS.
Plus près de chez nous, pourquoi devons-nous décrire ces choses clairement ?En fait, il s'agit aussi de vérifier la qualité de l'extraction de l'ARN.L'utilisation d'instruments et de kits rendra l'extraction d'ARN plus cohérente.L'échelle d'extraction des laboratoires ordinaires n'est pas grande et peut être obtenue avec des kits.
Les détails du traitement DNase ou RNase
Le problème important de la PCR quantitative fluorescente est d'empêcher la contamination de l'ADN et de ne pas expérimenter s'il y a contamination.Par conséquent, il est impératif d'indiquer le processus que vous avez utilisé pour traiter l'ADN, afin de démontrer que l'ADN dans le processus expérimental a été complètement et complètement supprimé.représenté par un schéma de principe.
Diagramme schématique de l'ARN et de l'ADN
En général, la méthode d'élimination de l'ADN consiste à traiter l'ARN avec de la DNase après extraction.Cependant, ce sont des méthodes relativement anciennes.Les kits d'extraction d'ARN commerciaux ont pu éliminer l'ADN pendant le processus d'extraction sans ajouter de DNase.Par exemple, une série de kits de Foregene .
Note: L'élimination de l'ADN lors de l'extraction de l'ARN est une épée à double tranchant très dangereuse, qui prolongera le temps de fonctionnement de l'extraction de l'ARN et augmentera le risque de dégradation de l'ARN.Fondamentalement, il s'agit d'un compromis entre le rendement et la pureté de l'ARN.
De plus, la quantité de DNase ajoutée à la colonne d'adsorption à base de silice est très faible et une DNase de haute qualité doit être utilisée pour obtenir l'effet.La DNase non optimisée ne peut pas être digérée rapidement et complètement.Il s'agit d'un test du niveau technique du commerçant.Bien sûr, il y a encore plus de marchands étranges qui se vantent que l'ADN peut être retiré sans DNase.On peut dire que quiconque se vante que l'ADN peut être complètement éliminé sans DNase est un hooligan.L'ADN est une structure bicaténaire relativement stable, et il ne peut pas être effacé simplement en parlant et en riant.
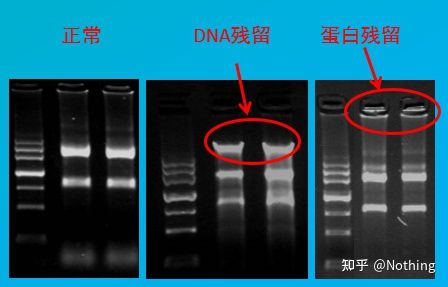
Évaluation de la contamination
méthode d'évaluation : détection par électrophorèse, 1 % d'agarose, 6 V/cm, 15 min, chargement 1-3 ul
Analyse quantitative des acides nucléiques
est généralement mesurée à l'aide d'un spectrophotomètre UV.Permettez-moi d'abord de vulgariser la signification des trois valeurs de OD260, OD280 et OD230.
·OD260nm : C'est la longueur d'onde d'absorption du pic d'absorption le plus élevé de l'acide nucléique, et la meilleure valeur mesurée varie de 0,1 à 1,0.Si ce n'est pas le cas, diluez ou concentrez l'échantillon pour le ramener dans la plage.
·OD280nm : C'est la longueur d'onde d'absorption du pic d'absorption le plus élevé des protéines et des substances phénoliques.
·OD230nm : C'est la longueur d'onde d'absorption du pic d'absorption le plus élevé des glucides.
Parlons ensuite du rôle de chaque indicateur.Pour A260, il peut être utilisé pour mesurer le rendement en acide nucléique.Lorsque OD260=1, dsDNA=50μg/ml, ssDNA=37μg/ml, RNA=40μg/ml.
Pour la pureté, nous devons regarder les ratios que nous voyons couramment : OD260/280 et OD260/230.
·ADN pur : OD260/280 est approximativement égal à 1,8.Lorsqu'il est supérieur à 1,9, cela indique qu'il y a une pollution par les ARN, et lorsqu'il est inférieur à 1,6, cela indique qu'il y a une pollution par les protéines et les phénols.
·ARN pur : 1,7
·OD260/230 : Qu'il s'agisse d'ADN ou d'ARN, la valeur de référence est de 2,5.Lorsqu'il est inférieur à 2,0, cela indique qu'il y a pollution par le sucre, le sel et la matière organique.
Intégrité de l'ARN
Il est très important de mesurer l'intégrité de l'ARN.Généralement, il est nécessaire de faire une expérience de gel de dénaturation d'ARN pour vérifier si la luminosité entre l'ARN 28S et 18S est une relation double.Lorsque la troisième bande 5S apparaît, cela signifie que l'ARN a commencé à se dégrader, sauf pour les invertébrés.
Données pour l'évaluation de la qualité de l'ARN : outre les tests ci-dessus, il existe également des tests d'instruments plus avancés en termes d'intégrité de l'ARN, tels que le test d'intégrité RQI du système d'électrophorèse automatique Experion, qui peut détecter si l'ARN est dégradé de manière invisible.
Dans la recherche scientifique, la PCR quantitative fluorescente est une comparaison entre le gène cible et le gène de référence interne.Par conséquent, dans le processus de conservation des échantillons d'ARN, d'extraction d'ARN, etc., l'objectif principal est d'assurer l'intégrité de l'ARN.
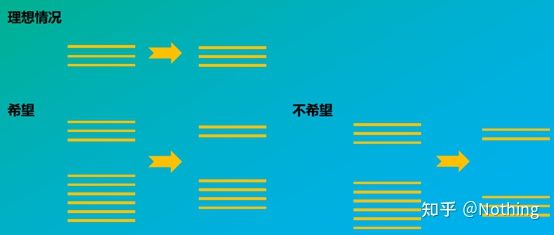
La manière dont l'intégrité de l'ARN affecte l'équilibre entre le gène cible et le gène de référence interne peut être facilement comprise à partir de la figure ci-dessous.La dégradation conduira à l'incomplétude du gène, qu'il s'agisse de l'incomplétude du gène de référence interne ou de l'incomplétude du gène cible, elle aura un impact important sur les données.
Diagramme schématique du gène cible et du gène de référence, ne doit pas être vrai
Test d'inhibition (si la valeur CT est supprimée sous une concentration élevée ou faible ou dans d'autres conditions)
En prenant cette figure comme exemple, les valeurs Ct des cinq courbes sont les suivantes.La distribution des valeurs de CT entre les courbes est inégale et les valeurs de Ct sont retardées sous des concentrations élevées et faibles, ce qui est le cas de l'inhibition de la PCR.
Point clé : dans le processus d'extraction d'ARN, nous devons abandonner les idées fausses et en établir de bonnes.
L'idée fausse est la suivante : l'extraction d'ARN ne fait que poursuivre le rendement, en pensant que plus la quantité d'ARN obtenue est importante, mieux c'est.En fait, quand on fait de la quantification, si le nombre de gènes n'est pas très grand, on n'a pas besoin de beaucoup d'ARN.La quantité d'ARN que vous extrayez est plus que suffisante.
La bonne notion est :L'extraction d'ARN doit rechercher la pureté, l'intégrité et la cohérence.La pureté peut garantir que la transcription inverse ultérieure n'est pas inhibée et que les données ne seront pas affectées par l'ADN.L'intégrité assure l'équilibre des séquences cibles et des références internes.La cohérence assure un chargement stable de l'échantillon.
MIQE (4) – transcription inversée
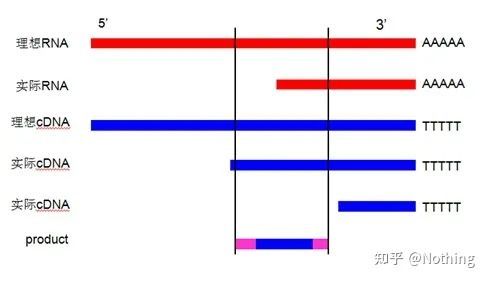
Idée fausse: la poursuite d'un volume d'échantillon plus élevé.
Notion correcte: Poursuivre la cohérence (stabilité), quelle que soit la quantité d'ARN chargé, l'efficacité de la transcription inverse reste constante, garantissant que les différences d'ADNc peuvent véritablement refléter les différences d'ARNm.
Nous expliquons ce processus avec un diagramme schématique :
Schéma de principe de l'efficacité de la transcription inverse, ne soyez pas vrai
Tout d'abord, nous devons comprendre la différence entre le processus de transcription inverse et le processus de PCR.La PCR subit de multiples processus de chauffage et de recuit, et le fragment cible croît de façon exponentielle ;alors que la transcription inverse n'a pas ce processus, nous pouvons imaginer que la transcription inverse est en fait un à un Pendant le processus de réplication, autant de morceaux d'ARN
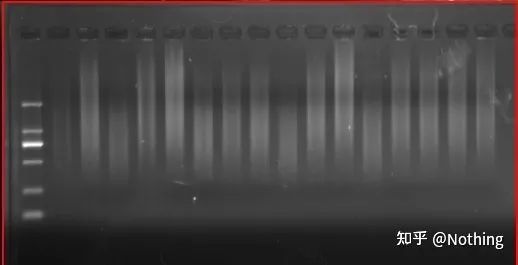
comme il peut y avoir autant d'informations sur l'ADNc, il faut le comprendre maintenant, car les fragments grands et petits ont été rétrotranscrits, et il est impossible de se concentrer sur un fragment.Et parce que la quantité d'ARN est relativement faible, la quantité d'ADNc obtenue est également relativement faible, contrairement à la PCR, qui a un effet d'amplification, il est donc fondamentalement impossible à détecter.
résultats d'électrophorèse d'ADNc
Deuxièmement, idéalement, la transcription inverse est effectuée un à un, mais aucune transcriptase inverse d'aucune entreprise ne peut obtenir cet effet.Fondamentalement, l'efficacité de la plupart des transcriptases inverses oscille entre 30 et 50 %.Si tel est le cas, nous aurions plutôt une efficacité de transcription inverse relativement stable, ce que nous voulons voir sur la figure : 3 ARN obtiennent 2 ADNc, 6 ARN obtiennent 4 ADNc, donc quelle que soit la quantité d'échantillon chargée, l'efficacité de la transcription inverse est relativement stable.Nous ne voulons pas voir la situation où l'efficacité de la transcription inverse est instable et une concentration élevée est inhibée.
Alors, comment vérifier si l'efficacité de la reverse transcription est stable ?La méthode est très simple, il vous suffit de faire un test de comparaison : l'un consiste à faire une transcription inverse en ADNc après avoir doublé la dilution de l'ARN, et l'autre consiste à faire une dilution double après la transcription inverse en ADNc, puis à faire une qPCR pour voir la pente obtenue est-elle cohérente.En tant que meilleur étudiant, vous devriez le comprendre en quelques secondes.Comme indiqué ci-dessous:
Dilution de l'ARN et de l'ADNc pour tester si l'efficacité de la transcription inverse est stable
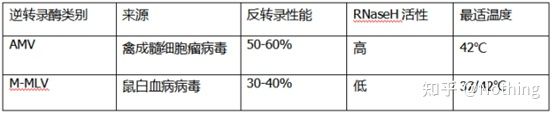
Transcriptase inverse et kit
Comment la PCR quantitative fluorescente parfaite peut-elle avoir une excellente transcriptase inverse et un kit.La transcriptase inverse est grossièrement divisée en deux types selon la source, AMV ouM-MLV, et leurs performances sont identiques à celles indiquées dans le tableau.
Activité RNase H
La RNase H est la ribonucléase H, le nom chinois est la ribonucléase H, qui est une endoribonucléase qui peut spécifiquement hydrolyser l'ARN dans la chaîne hybride ADN-ARN.La RNase H ne peut pas hydrolyser les liaisons phosphodiester dans l'ADN ou l'ARN simple brin ou double brin, c'est-à-dire qu'elle ne peut pas digérer l'ADN ou l'ARN simple brin ou double brin.Couramment utilisé dans la synthèse du deuxième brin d'ADNc.
C'est une chose étrange.Nous disons que la transcriptase inverse a une activité RNase H, et non que la transcriptase inverse contient de la RNase H, et il peut ne pas être possible de séparer la RNase H de la transcriptase inverse, peut-être à cause de la conformation de certains groupes dans la transcriptase inverse. Cette activité est causée par la transcriptase inverse.
Par conséquent, quelle que soit l'efficacité de transcription inverse plus élevée de l'AMV, son activité RNase H réduit le rendement en ADNc.Bien sûr, les fabricants de réactifs optimisent constamment leurs produits pour éliminer autant que possible l'activité RNase H dans la transcriptase inverse afin d'augmenter le rendement en ADNc.
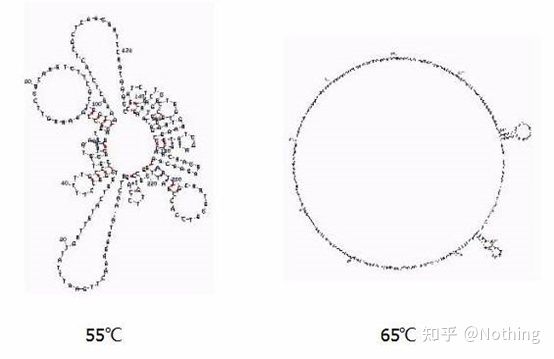
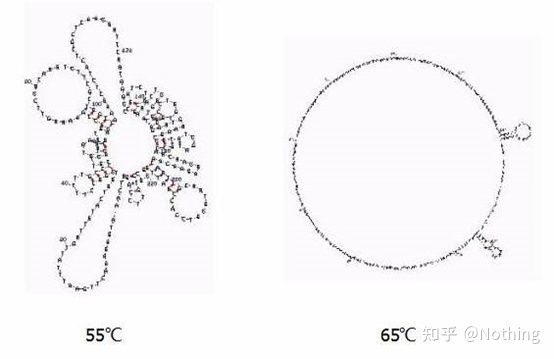
Température de recuit
Structure secondaire de l'ARN à différentes températures
Voir la figure ci-dessus pour la structure secondaire de l'ARN à différentes températures et utiliser l'outil en ligne mFold pour déterminer la structure secondaire du fragment cible dans des conditions de température et de concentration de sel spécifiques.A 55°C, la structure secondaire de l'ARN est encore très complexe, la transcriptase inverse ne peut pas fonctionner, et la structure secondaire ne peut être complètement résolue qu'à 65°C, alors que la température optimale de l'AMV et du M-MLV est bien inférieure à cette température.
ce qu'il faut faire?La structure secondaire est l'appariement complémentaire de la matrice elle-même, ce qui conduit à une forte compétition entre l'amorce et la transcriptase inverse et la matrice, entraînant une série de problèmes tels qu'un faible E et une mauvaise répétabilité.
ce qu'il faut faire?N'augmentez que la température de recuit autant que possible.
De nombreux fabricants de réactifs améliorent leur transcriptase inverse grâce au génie génétique.Certains augmentent la température de réaction, tels que Jifan et Aidelai, et certains éliminent le groupe actif de l'enzyme RNase H pour améliorer l'affinité entre l'enzyme et la matrice d'ARN.Une affinité élevée peut extraire de manière compétitive la structure secondaire et lire en douceur, et également améliorer considérablement l'efficacité de la transcription inverse.
Point clé : la transcription inverse est plus importante pour poursuivre la cohérence de l'efficacité de la transcription inverse (les enzymes doivent non seulement être efficaces mais également stables), plutôt que la quantité d'échantillon chargée, s'il ne s'agit pas d'une PCR quantitative fluorescente particulièrement à grande échelle, cela ne sera pas possible du tout.ADNc multiples.
Divers fabricants ont également fait des efforts dans la poursuite de la cohérence.Par exemple, la plupart des entreprises proposent désormais la transcription inverse sous forme de kit standard à vendre, ce qui est un bon choix.
Par exemple, les kits de la série RT Easy de Foregene :
RT Easy I (prémélange maître pour kit de synthèse d'ADNc premier brin)
MIQE (5) – informations sur les gènes cibles
La figure ci-dessus explique
1. L'efficacité de ce gène pour des expériences répétées peut généralement être vérifiée par des expériences répétées.
2. Gene ID, vous savez.
3. La longueur du gène, la longueur totale du gène cible n'est certainement pas un problème.Lors de la conception des amorces, assurez-vous que la longueur de l'amplicon est comprise entre 80 et 200 pb pour assurer une meilleure efficacité d'amplification.
4. Informations de comparaison Sequence Blast, le gène cible doit être comparé dans la banque de gènes pour éviter une amplification non spécifique.
5. Présence de pseudogènes.Un pseudogène est une séquence d'ADN similaire à un gène normal mais qui perd sa fonction normale.Il existe souvent dans la famille multigénique des eucaryotes.Il est généralement représenté par ψ.Il s'agit d'une copie d'ADN génomique non fonctionnelle dans le génome qui est très similaire à la séquence du gène codant., ne sont généralement pas transcrits et n'ont pas de signification physiologique claire.
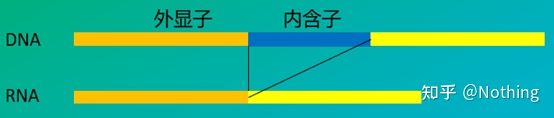
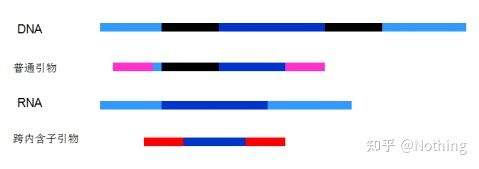
6. Position des amorces par rapport aux exons et aux introns.Dans les premières années, lorsque nous avons résolu le problème de la contamination de l'ADN, nous avons souvent prêté attention aux positions des amorces, des exons et des introns, et avons généralement envisagé de concevoir des amorces à travers les introns pour éviter l'amplification de l'ADN.Veuillez consulter la figure ci-dessous : le noir représente les introns, divers bleus représentent les exons, le rose représente les amorces communes et le rouge vif représente les amorces couvrant les introns.
Schématique, jamais vrai
Quel plan parfait cela semble-t-il, mais en fait, dans la plupart des cas, les amorces trans-intron ne sont pas aussi magiques qu'on l'imagine, et elles provoqueront également une amplification non spécifique.Ainsi, la meilleure façon de prévenir la contamination par l'ADN est de supprimer complètement l'ADN.
7. Prédiction de conformation.En utilisant à nouveau cet exemple, utilisez l'outil en ligne mFold pour déterminer la structure secondaire du fragment cible à une température et une concentration de sel spécifiques.
Structure secondaire de l'ARN à différentes températures
La structure secondaire est l'appariement complémentaire du modèle lui-même, ce qui conduira à une forte concurrence entre l'appariement de l'amorce et du modèle, et les chances de liaison de l'amorce sont moindres, ce qui entraîne une série de problèmes tels qu'un faible E et une mauvaise répétabilité.Grâce à la prédiction logicielle, s'il n'y a pas de problème de structure secondaire, ce serait formidable.Si c'est le cas, notre article de suivi discutera spécifiquement de la façon de résoudre ce problème.
MIQE (6)—Oligonucléotides qPCR
Pour la PCR quantitative fluorescente, la première chose avec laquelle vous luttez chaque jour est l'extraction d'ARN, et la deuxième chose peut être la conception d'amorces.
Tout d'abord, nous vérifions toujours les règles de conception des amorces selon la liste de contrôle MIQE.C'est si simple que les scumbags peuvent rire, et nous pouvons le terminer en une phrase : découvrez la séquence et la position de la sonde d'amorce et la méthode de modification.Pour la méthode de purification des amorces, la synthèse des amorces est si bon marché à l'heure actuelle, la qPCR est digne des méthodes de purification PAGE et supérieures, et les informations de l'instrument de synthèse ne sont pas importantes.Beaucoup de gens font des amorces depuis des décennies et ne savent pas que le synthétiseur est ABI3900.
En ce qui concerne les principes de conception d'amorces, vous n'avez pas à les mémoriser par cœur, car la plupart des logiciels de conception d'amorces ou des outils en ligne peuvent prendre en charge ces problèmes (outil en ligne recommandé primer3.ut.ee/), et 99,999 % de la conception d'amorces ne se fait pas manuellement. Regardez, l'auteur conçoit parfois des centaines d'amorces par jour, si vous les lisez une par une, cela deviendra louche.
Vérifiez simplement les points suivants après la conception des amorces :
1. Concevoir des amorces proches de l'extrémité 3' : Dans le cas de l'utilisation d'amorces oligo dT pour la synthèse du premier brin d'ADNc, compte tenu de l'efficacité de la transcription inverse et de l'intégrité de l'ARN, les amorces conçues doivent être conçues près de l'extrémité 3' pour améliorer l'efficacité de l'amplification .Utilisez une image pour expliquer comme suit (il n'y a aucun moyen de comprendre cela):
Pourquoi les amorces devraient-elles être conçues près de l'extrémité 3 ', cela ne doit pas être vrai
2. Valeur TM : La valeur Tm est à 55-65°C (parce que l'activité exonucléase est la plus élevée à 60°C), et la teneur en GC est à 40%-60%.
3. BLAST : Afin d'éviter une amplification non spécifique du génome, Blast doit être utilisé pour une vérification supplémentaire.
MIQE(7)—processus de qPCR
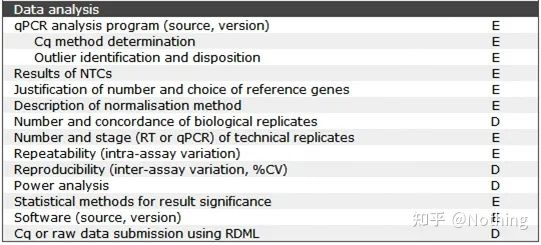
1. Trousse qPCR
Selon les exigences du MIQE, nous devons clairement décrire les conditions de réaction complètes dans l'article, y compris la configuration du système de réaction PCR, quel kit est utilisé, qui est le fabricant, quelle est la taille du système de réaction, si la méthode du colorant ou la méthode de la sonde est utilisée, les paramètres du programme PCR.Les conducteurs vétérans trouveront certainement que tant que le kit est sélectionné, les informations ci-dessus sont essentiellement déterminées.
À l'heure actuelle, la fabrication et la production de kits PCR quantitatifs fluorescents est une technologie très mature.Tant que vous ne choisissez pas des fabricants extrêmement mauvais, la probabilité de problèmes n'est pas élevée, mais nous souhaitons tout de même partager avec vous quelques points :
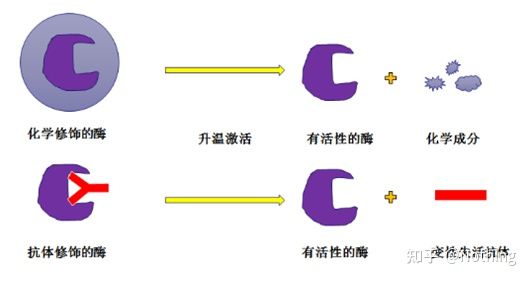
Enzyme Taq à démarrage à chaud :La partie la plus importante de la PCR est l'enzyme Taq à démarrage à chaud.Les enzymes de démarrage à chaud sur le marché sont généralement divisées en deux types, l'une est une enzyme de démarrage à chaud chimiquement modifiée (vous pouvez l'imaginer comme une incorporation de paraffine), et l'autre est une enzyme de démarrage à chaud pour la modification d'anticorps (liaison antigène-anticorps).La modification chimique est un moyen précoce d'enzymes de démarrage à chaud.Lorsqu'une certaine température est atteinte, l'enzyme libère son activité.L'enzyme de démarrage à chaud modifiée par un anticorps utilise des méthodes biologiques pour bloquer l'activité de l'enzyme.Lorsqu'une certaine température sera atteinte, l'anticorps sera dénaturé et inactivé en tant que protéine, et l'activité enzymatique sera mise en jeu.
Cependant, à quoi cela sert-il ?C'est le cas, l'activité de libération des enzymes modifiées par anticorps est plus rapide que celle des enzymes modifiées chimiquement, donc en termes de sensibilité, les enzymes modifiées par anticorps ont un léger avantage, de sorte qu'il n'y a pratiquement pas d'enzymes modifiées chimiquement dans les kits sur le marché.Si c'est le cas, alors la technologie de ce fabricant est encore coincée dans l'ère du millénaire.
Concentration en ions magnésium :La concentration en ions magnésium est très importante dans la réaction PCR.Une concentration appropriée en ions magnésium peut favoriser la libération de l'activité de l'enzyme Taq.Si la concentration est trop faible, l'activité enzymatique sera significativement réduite ;si la concentration est trop élevée, l'amplification non spécifique catalysée par l'enzyme sera renforcée.La concentration d'ions magnésium affectera également l'hybridation des amorces, la température de fusion de la matrice et des produits de PCR, affectant ainsi le rendement en fragments amplifiés.La concentration des ions magnésium est généralement contrôlée à 25 mM.Bien sûr, pour un bon kit, la concentration en ions magnésium doit être bien contrôlée.Certains marchands ajoutent un agent chélatant les ions magnésium au réactif, ce qui peut obtenir l'effet d'ajustement automatique de la concentration en ions magnésium.
Concentration de colorant fluorescent :Le colorant fluorescent, qui est le SYBR Green que nous utilisons habituellement, génère principalement de la fluorescence en se liant au petit sillon de l'ADN double brin, car la liaison du colorant à l'ADN double brin est non spécifique, c'est-à-dire Tant que l'ADN double brin est combiné avec lui, une fluorescence peut se produire, de sorte que les dimères d'amorces et les matrices d'ADN du système se combinent avec lui pour former un signal de fond.
PS : En raison de ses propriétés photosensibles, les produits du marché sont généralement conditionnés dans des tubes à centrifuger marron opaques (comme le montre l'image ci-dessous).Cependant, cela rencontrera un problème.Il est difficile de voir si le liquide est aspiré lors du prélèvement.À cet égard, Qingke est en effet le plus convivial (comme le montre l'image ci-dessous), et le tube transparent est emballé dans un sac en étain opaque.Ensuite, mettez-le dans un sac en étain, en tenant compte de la commodité d'éviter la lumière et l'échantillonnage.Vous devez choisir le bon numéro de produit.TSE204 est une existence super rentable, ce qui me donne envie de planter de l'herbe.
La concentration du colorant fluorescent est également très importante.Si la concentration est trop faible, la courbe d'amplification ne montera pas dans l'étape ultérieure et n'est pas parfaite ;si la concentration est trop élevée, cela provoquera des interférences sonores.Étant donné que la PCR quantitative fluorescente dépend principalement de la valeur CT, si la concentration du colorant fluorescent n'est pas ajustée correctement, le point bas est meilleur que le point haut.Bien sûr, la concentration de colorant appropriée est la meilleure.
ROX: Les colorants ROX sont utilisés pour corriger les erreurs de signal de fluorescence de puits à puits.Certains fabricants d'instruments exigent un étalonnage, d'autres non.Par exemple, l'utilisation de l'instrument d'amplification PCR en temps réel de Thermo Fisher Scientific nécessite généralement un étalonnage, y compris 7300, 7500, 7500Fast, StepOnePlus, etc. Les instructions générales du kit le décriront.
Le mélange qPCR de Foregene contient également un colorant ROX, qui est pratique pour une utilisation dans divers modèles.
Kit PCR en temps réel - Taqman
Traitement des liaisons hydrogène faibles: Le traitement des liaisons hydrogène faibles est un sujet relativement technique.Rien n'a lu les manuels de nombreux kits, mais aucun d'entre eux n'a mentionné ce sujet.En fait, c'est tellement important.La combinaison de bases dépend principalement de la force des liaisons hydrogène.Les liaisons hydrogène fortes sont une amplification normale et les liaisons hydrogène faibles conduisent à une amplification non spécifique.Si les liaisons hydrogène faibles ne peuvent pas être bien éliminées, une amplification non spécifique ne peut être évitée.Dans le cadre de l'auteur, seules quelques entreprises ont remarqué ce problème.Lorsque vous achetez le kit, vous pouvez indiquer si vous avez envisagé une solution à cet égard pour le kit que vous souhaitez choisir.
Volume de réaction: Le système 20-50ul est plus couramment utilisé, et des volumes plus petits sont susceptibles de provoquer des erreurs.D'une manière générale, les instructions du kit recommanderont l'utilisation de volumes de réaction PCR.Ne soyez pas intelligent et utilisez de plus petits volumes pour réduire les coûts.l'objectif de.Le volume recommandé par les marchands a effectivement été testé, et il se peut qu'ils ne puissent pas résoudre le problème des erreurs causées par les petits volumes.
2. Le fabricant et le numéro d'article de la plaque tubulaire
Tout le monde connaît le principe de la PCR quantitative fluorescente.La collecte de fluorescence est principalement réalisée à travers des bouchons de tubes PCR.Lors du choix des consommables PCR, faites attention à deux points : une bonne transmission de la lumière et une adaptation à l'instrument.D'une manière générale, les platines et tubes des marques grand public sont bien, mais il faut bien choisir en terme d'adaptation, sinon on ne pourra pas utiliser l'instrument.
4. Connaissances de haut niveau
MIQE (8)—validation qPCR
C'est la priorité absolue de la qPCR !Tant de héros sont tombés dans le sable ici.Bien sûr, il est également possible que vous ayez de la chance et que les gènes que vous avez étudiés soient simples, vous avez donc flotté dans la grotte de glace au gré du vent.Les informations de vérification de qPCR sont destinées à tester la fiabilité des données.Nous listons les informations de vérification nécessaires comme suit :
1. Test de spécificité
La spécificité de l'amplification du gène cible est testée en vérifiant si l'image d'électrophorèse est une bande unique ;vérification du séquençage ;courbe de fusion pour voir si la carte de pic est unique ;vérification de la digestion enzymatique et autres méthodes.
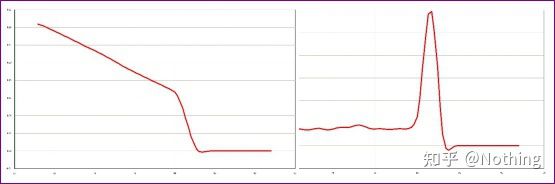
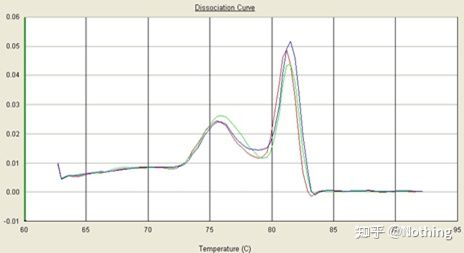
Ici, nous nous concentrons sur tl'analyse de l'amplification non spécifique par la méthode des courbes de fusion.D'une manière générale, lorsque nous concevons des amorces, la taille du fragment de produit doit être comprise entre 80 et 200 pb, ce qui rend la température de fusion du produit de PCR comprise entre 80 et 85 °C.Par conséquent, s'il existe des pics divers, il doit y avoir d'autres produits d'amplification non spécifiques ;si le pic apparaît en dessous de 80°C, il est généralement considéré comme un dimère d'amorce ;si le pic apparaît au-dessus de 85°C, on considère généralement qu'il s'agit d'une contamination de l'ADN ou d'une amplification non spécifique de gros fragments.
Remarque : Parfois, il n'y a qu'un seul pic à 80 °C.À l'heure actuelle, ce concept doit être respecté.Il est probable que les résultats d'amplification soient tous des dimères d'amorces.
Courbe de fusion normale (pic unique sans amplification non spécifique)
Courbe de fusion problématique (amplification non spécifique des pics parasites)
【Analyse de cas】
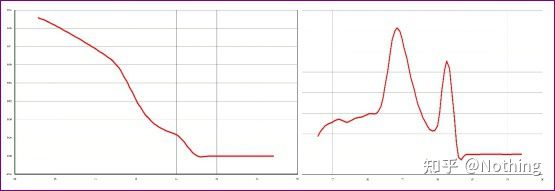
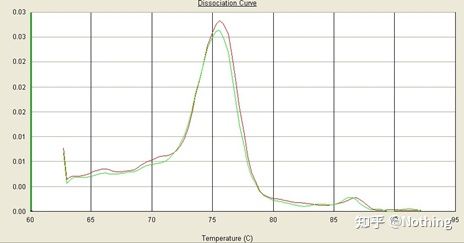
Il y a un pic principal, mais le dimère d'amorce est sérieux
La courbe de fusion à pic unique dans la figure ci-dessous peut facilement tromper vos yeux, pensant que c'est une expérience parfaite, mais le résultat est complètement faux.A ce moment, nous devons regarder la température de fusion.La température maximale est inférieure à 80 ° C, ce qui est complètement dimère d'amorce.
Aucun fragment cible, tous les dimères d'amorce
Ici, mon frère ne peut pas s'arrêter.La photo ci-dessous est une photo prise avec un téléphone portable qui m'a été envoyée par un salaud.Les réactifs qu'il a utilisés sont tous des marques couramment utilisées dans l'industrie.Il est passé d'une marque de préfixe T à une autre marque de préfixe T.Je pense que vous l'avez déjà deviné.Le salaud m'a crié : « Le réactif utilisé sur la première photo est trop bon, et le pic est unique.Plus tard, après avoir utilisé le réactif que vous avez recommandé, il devient comme la deuxième image, avec des pics mixtes.Vous m'avez rendu malheureux."
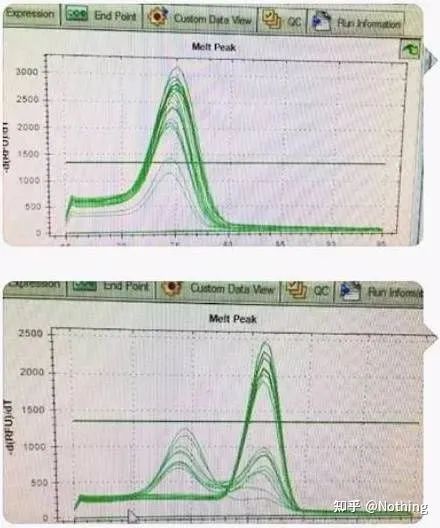
Séparez les deux graphiques.À première vue, l'un a un seul pic et l'autre un double pic.Non-sens, un seul pic est bien sûr très bien.Est-ce vrai?
Pire que Dou E, si je mets les deux photos dans l'image ci-dessous, vous comprendrez tout de suite.En fait, on est facilement paralysé par ce genre d'image.Après une analyse minutieuse, nous avons trouvé que : le pic du premier chiffre est à 75°C, ce qui est complètement un dimère d'amorce ;le pic du deuxième chiffre apparaît à 75°C et 82°C, au moins il y a Le produit apparaît.
Photos de retours d'élèves
Ainsi, le problème fondamental n'est pas le problème des réactifs, mais le problème de la conception des amorces.En même temps, ça prouve aussi que certaines grandes marques ne sont pas de qualité fer, et ça prouve aussi ce que disait mon frère avant : Ce n'est pas la marque de réactif qui supporte ton article.C'est votre article qui a étayé la marque de réactifs.Imaginez, si le connard ne changeait pas les réactifs, les mauvaises données seraient envoyées au journal, et ce qui se passerait serait une tragédie.
2. Valeur Ct du contrôle à blanc
N'expliquez pas, si le contrôle à blanc a une valeur Ct, n'est-ce pas une pollution ?Cependant, vous devez toujours comprendre quel contrôle à blanc a une valeur Ct.S'il s'agit de NTC, cela signifie qu'il y a de l'ADN étranger tel qu'une contamination par un réactif.S'il s'agit de NRT, cela signifie que l'ARN extrait est contaminé par l'ADN.
3. Courbe standard
Y compris la pente et la formule de calcul, l'efficacité de la PCR peut être calculée à l'aide de la formule.Une expérience parfaite nécessite que la pente de la courbe standard approche 3,32 et que R² approche 0,9999.
4. Plage dynamique linéaire
La plage dynamique de la réaction est linéaire.Selon le modèle utilisé pour générer la courbe standard, la plage dynamique doit inclure au moins 5 gradients de concentration et prêter attention au changement des valeurs Ct à des gradients de concentration élevés et à des gradients de faible concentration.
5. Précision de détection
Les changements dans les résultats de qPCR, c'est-à-dire une mauvaise répétabilité, c'est-à-dire une mauvaise précision, sont causés par de nombreux facteurs, notamment la température, la concentration et le fonctionnement.La précision de la qPCR devient généralement moins contrôlable à mesure que le nombre de copies diminue.Idéalement, la variation intra-expérimentale, cette variation technique doit être distincte de la variation biologique, et les répliques biologiques peuvent traiter directement les différences statistiques dans les résultats de la qPCR entre les groupes ou les traitements.En particulier pour les tests de diagnostic, la meilleure précision inter-test (répétabilité) entre les sites et les opérateurs doit être signalée.
6. Efficacité de détection et LOD (en multiplex qPCR)
LOD est la plus faible concentration de 95 % d'échantillons positifs détectés.En d'autres termes, la concentration de LOD contenue dans un ensemble de réplicats de gènes cibles ne doit pas dépasser 5 % des réactions ayant échoué.Lors de l'analyse qPCR multiplex, en particulier pour la détection simultanée de mutations ponctuelles ou de polymorphismes, la qPCR multiplex doit fournir la preuve que la précision de plusieurs fragments cibles n'est pas compromise dans le même tube, l'efficacité et la LOD de détection multiple et de détection de tube unique doivent être les mêmes.En particulier lorsque des gènes cibles à haute concentration et des gènes cibles à faible concentration sont amplifiés simultanément, ce problème doit être pris en compte.
Problèmes et solutionsD'une manière générale, les problèmes souvent rencontrés dans le débogage qPCR portent sur les aspects suivants :
·amplification non spécifique
·Choix difficile de la concentration d'amorce et problème avec les amorces-dimères
·La température de recuit est imprécise
·La structure secondaire affecte l'efficacité de l'amplification
amplification non spécifique
amplification non spécifiquese produit, on considère généralement si la conception de l'amorce ne convient pas, mais si vous n'êtes pas pressé de changer les amorces, vous pouvez d'abord essayer les méthodes suivantes (le principe est également joint):
·Augmentez la température de recuit - essayez de rendre les liaisons hydrogène faibles incapables de se maintenir ;
· Raccourcir le temps de recuit et d'allongement - réduire le risque de liaisons hydrogène faibles ;
·Réduire la concentration d'amorces - réduire le risque de liaison d'amorces redondantes et de régions non cibles ;
Faible efficacité d'amplification
La situation opposée à l'amplification non spécifique - faible efficacité d'amplification , et les mesures pour faire face à une faible efficacité d'amplification sont tout le contraire :
·Prolonger le temps de recuit et d'allongement ;
·Passez à la PCR en trois étapes et réduisez la température de recuit ;
·Augmenter la concentration de l'apprêt ;
Ps: De nombreux étudiants diplômés nés dans les années 90 ne sont pas disposés à étudier comment déboguer des expériences et espèrent que le kit pourra résoudre complètement le problème (si vous souhaitez vous rendre dans une société de réactifs pour faire de la recherche et du développement après l'obtention de votre diplôme), en fait, les fabricants de réactifs pensent également de cette façon, j'espère que c'est un imbécile Il peut être utilisé lorsque vous l'obtenez, donc les fabricants de réactifs ont déployé beaucoup d'efforts pour résoudre le problème de l'amplification non spécifique, y compris l'introduction de facteurs d'absorption de liaison H faibles.Pour résoudre facilement le problème, les imbéciles doivent encore lire l'introduction de la société de réactifs pour voir s'il existe un facteur qui absorbe les liaisons hydrogène faibles.
Choix difficile de la concentration des amorces et problème avec les amorces-dimères
Méthode 1: D'une manière générale, les instructions du kit pour qPCR ont des systèmes recommandés et des concentrations d'amorces recommandées.
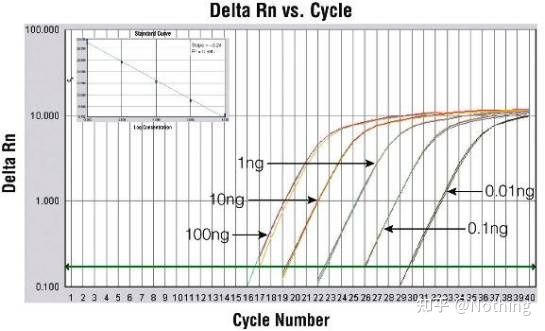
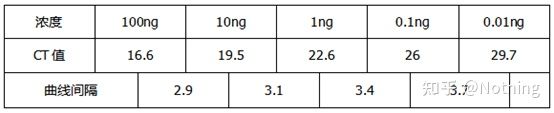
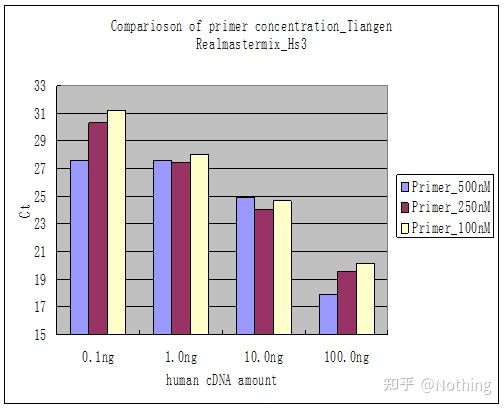
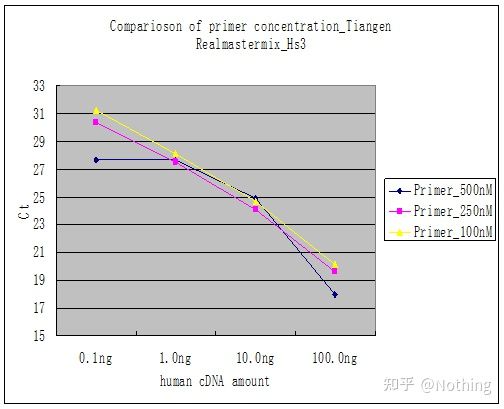
Méthode 2: Débogage en définissant le gradient de concentration d'amorce.La photo ci-dessous est volée à une entreprise pour illustrer.La figure ci-dessous montre les résultats quantitatifs de fluorescence obtenus avec trois gradients de concentration d'amorce (100 nM, 250 nM, 500 nM) et quatre gradients de concentration de matrice (0,1 ng, 1 ng, 10 ng, 100 ng).La valeur Ct des résultats expérimentaux est tracée comme suit :
Sélection de la concentration d'amorce Concaténez chaque concentration d'amorce en une ligne comme suit :
Le choix de la concentration d'amorce est évident, la relation linéaire de la concentration d'amorce de 100 nM et 250 nM est meilleure, et la relation linéaire de la concentration d'amorce de 500 nM est relativement mauvaise.À 100 nM et 250 nM, la valeur Ct de 250 nM est relativement faible, de sorte que la concentration optimale d'amorce est de 250 nM.Des amorces-dimères généralement sévères peuvent être observées dans la courbe de fusion.Que se passe-t-il si les amorces conçues ne peuvent pas éviter les amorces-dimères ?
Méthode 3: Réduire la quantité d'amorces et augmenter la température de recuit (pas besoin d'expliquer).
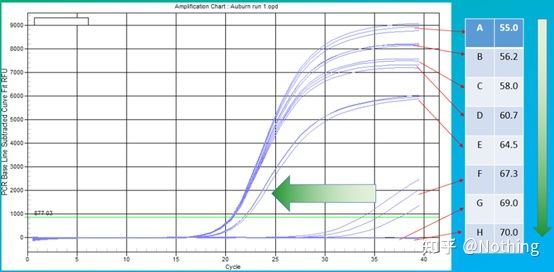
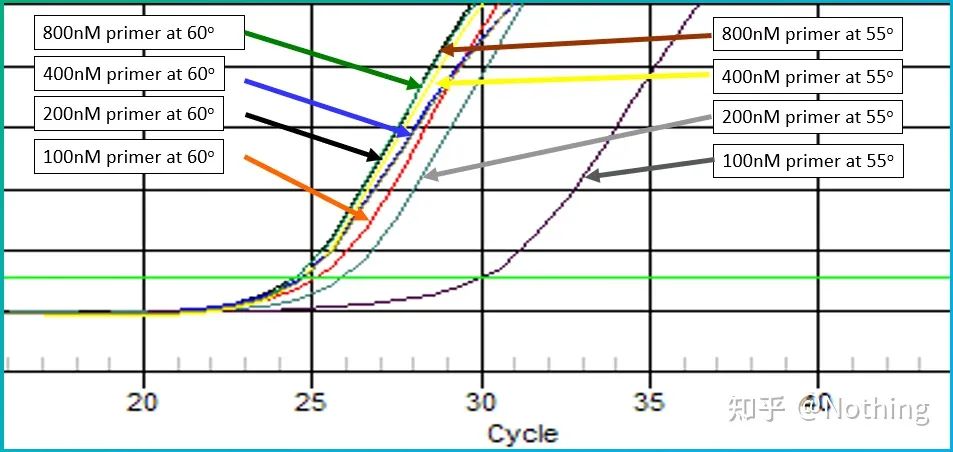
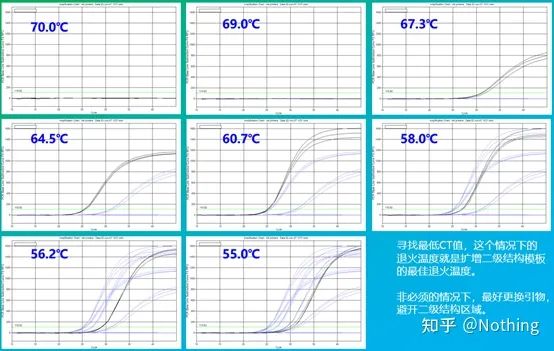
La valeur empirique de la température de recuit est de 60°C.Si vous n'êtes pas sûr, comment choisir une température de recuit plus appropriée ?La réponse est la même que le choix de la concentration d'amorce -essai de gradient.Prenez une photo de la société Bio-rad pour illustrer le problème.Pour l'amplification d'un certain fragment cible, définissez huit gradients de température, chacun avec trois répétitions, et la courbe d'amplification obtenue est la suivante :
sélection de la température de recuit :
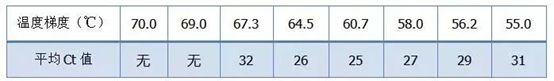
·70°C, 69°C— Fondamentalement, les amorces ne peuvent pas être combinées, il n'y a donc pas d'amplification.
·67,3°C – Il y a une petite quantité d'amplification au début, et la valeur Ct est relativement grande.
·64,5°C——La valeur Ct diminue.
·À 60,7 °C, 58,0 °C, 56,2 °C et 55,0 °C, les valeurs de Ct avaient fondamentalement tendance à être stables, mais les valeurs finales de fluorescence étaient différentes.
Comment choisir?Principe : Le premier principe est la valeur Ct plus élevée.Pour la même valeur Ct, choisissez une température de recuit plus élevée pour éviter la dimérisation et l'amplification non spécifique.Bien qu'il y ait une valeur de fluorescence plus élevée à 55°C, il peut y avoir des dimères ou une amplification non spécifique.
Mais si vous êtes aussi intelligent que vous, vous penserez certainement : Logiquement parlant, si la réaction PCR est très spécifique, tant que la concentration d'amorce dépasse l'exigence minimale, les points haut et bas ne devraient avoir aucun effet, tout comme les colorants fluorescents et les dNTP.En effet, tant que la température de recuit est optimisée correctement, l'effet de la concentration de l'amorce sur la valeur Ct sera naturellement minimisé.
La température de recuit est optimisée correctement et l'effet de la concentration d'amorce sur CT sera minimisé
La structure secondaire affecte l'efficacité de l'amplification
Prenons l'image de Bio-rad pour illustrer le problème.Il conçoit également un gradient de température pour amplifier un gène avec une structure secondaire.
La structure secondaire émerge
On peut voir qu'à mesure que le gradient de température diminue, les produits commencent à apparaître et la valeur Ct progresse, atteignant la valeur minimale à 60,7 ° C, puis à mesure que le gradient de température diminue, la valeur Ct devient plus grande.A l'inverse, lorsque la température augmente, la structure secondaire s'ouvre et le rendement d'amplification augmente.Après avoir atteint une certaine température, l'augmentation de la température ne peut pas améliorer l'efficacité de l'amplification.Parce que les amorces ne peuvent pas être combinées de manière stable à ce moment.Donc,rechercher la température avec la valeur Ct la plus faible, qui est la meilleure température pour amplifier le modèle de structure secondaire !Bien sûr, les imbéciles intelligents doivent savoir que si ce n'est pas nécessaire, il est préférable de changer les amorces et d'éviter la région de structure secondaire.
5. Niveau d'application
MIQE—Analyse des données
L'analyse des données est principalement donnée par l'instrument PCR quantitatif fluorescent.Dans l'article précédent, de nombreux travaux d'analyse de données ont été effectués, comme le contrôle à blanc, qui a été expliqué dans la conception de l'expérience.Les gènes de référence internes, les numéros de répétition, etc. ont été clarifiés., nous expliquons ici principalement l'application de la qPCR.
La qPCR est largement utilisée, et la vérification expérimentale et le diagnostic des acides nucléiques sont les scénarios les plus couramment utilisés.
quantification absolue
Log (concentration initiale) a une relation linéaire avec le nombre de cycles.Une courbe standard peut être tracée à partir d'un standard avec un nombre de copies initial connu, c'est-à-dire que la relation linéaire de la réaction d'amplification peut être obtenue.Selon la valeur Ct de l'échantillon, la concentration dans l'échantillon peut être calculée.Le nombre de modèles à inclure.
Méthode de calcul quantitatif absolu
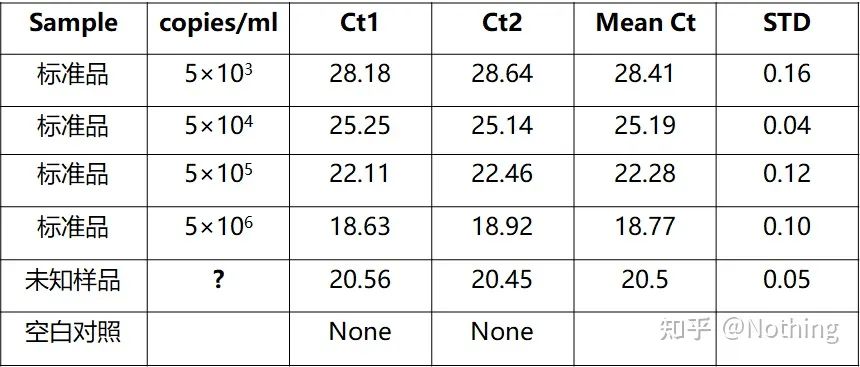
La quantification absolue doit être basée sur la courbe standard.Pour faire une courbe standard, un standard est nécessaire.Habituellement, le standard est un plasmide obtenu par clonage du gène cible.Pourquoi est-ce un plasmide ?Parce que l'ADN plasmidique circulaire est le plus stable.Diluer le produit standard en 5 à 6 gradients selon le rapport de doublement (dilution 10 fois), et faire attention à l'uniformité lors de la dilution.Laissez la valeur Ct se situer entre 15 et 30.
Préparation standard
Dans le même temps, l'échantillon à tester doit également être dilué en conséquence (rappelez-vous le facteur de dilution) et la valeur Ct doit également se situer entre 15 et 30.Le produit standard + l'échantillon à tester sont mis ensemble sur la machine.Après l'analyse, une courbe standard a été réalisée avec la substance standard et les échantillons à tester ont été amenés dans la courbe standard pour calculer la concentration.
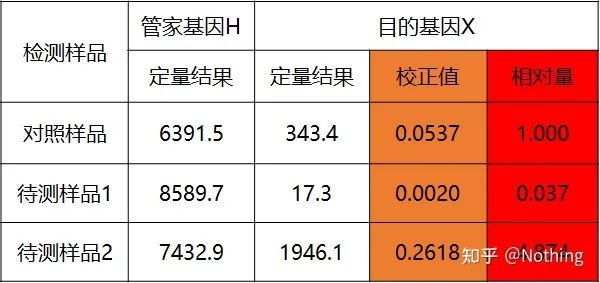
La quantification du VHB du virus de l'hépatite B est une quantification absolue typique, qui peut calculer le nombre de copies du virus dans 1 ml de sang.
Calcul du nombre de copies
Concentration de l'échantillon à tester (ng/ul) = OD260 × 50ug/ml × facteur de dilution
Poids moléculaire de l'échantillon = nombre de bases × 324
Le nombre de copies de l'échantillon à tester (copies/ul) = la concentration de l'échantillon à tester / le poids moléculaire de l'échantillon × 6 × 1014
Méthode de calcul du nombre de copies
Ce qui précède est la méthode de calcul pour déterminer la quantité.Il s'agit d'un problème mathématique qui peut être résolu après avoir obtenu son diplôme d'études secondaires, et les problèmes mathématiques sont généralement résolus par des ordinateurs.Si vous ne comprenez pas, vous pouvez venir communiquer.
quantification relative
La quantification relative est principalement utilisée dans la recherche scientifique.Combien de virus y a-t-il dans 1 ml de sang, et c'est un virus à ADN, c'est un événement relativement déterministe : la quantité de sang peut être déterminée, et le virus à ADN est relativement stable.Cependant, il nous est difficile de comparer le nombre de copies de transcription d'un certain gène dans une feuille, car il est difficile de déterminer la taille, le poids et la tendreté de la feuille, la quantité d'ARN extrait est difficile à déterminer, et l'efficacité de la transcription inverse est également difficile à déterminer, c'est-à-dire que toute étape peut rendre les données expérimentales erronées et inutilisables.
Par conséquent, la quantification relative doit introduire un élément :le gène de référence interne.
En d'autres termes, la quantification relative est en fait une comparaison entre le gène cible et le gène de référence interne.Par rapport au même tissu et à la même cellule, l'influence de la taille de l'échantillon, de la quantité d'extraction d'ARN, de l'efficacité de la transcription inverse et de l'efficacité de la PCR est relativement faible.En raison de la petite taille de l'échantillon, les gènes de référence internes et les gènes cibles ont été relativement réduits.C'est pourquoi nous avons mis l'accent sur l'uniformité et la stabilité auparavant.
Les gènes de référence internes sont généralementgènes de ménage(gènes domestiques), qui font référence à une classe de gènes qui sont exprimés de manière stable dans toutes les cellules, et leurs produits sont nécessaires pour maintenir les activités vitales de base des cellules.
Ne confondez pas ce concept.Les gènes domestiques sont des termes de fonction biologique, tandis que les gènes de référence internes sont des termes techniques expérimentaux.Les gènes domestiques doivent passer la validation avant de pouvoir être sélectionnés comme gènes de référence internes.
Par exemple, nous avons sélectionné plusieurs gènes domestiques dans la figure ci-dessous pour tester leurs niveaux d'expression dans différentes cellules tissulaires, et avons constaté que les niveaux d'expression de la β-2-microglobuline étaient assez différents de ceux des trois autres gènes, de sorte qu'ils ne pouvaient pas être utilisés comme gènes de référence internes.
Après avoir compris la fonction de correction du gène de référence interne, deux algorithmes sont dérivés en raison de l'introduction du gène de référence interne.
·méthode de la double courbe standard
·2 – Méthode △△Ct (méthode de comparaison des valeurs CT)
Si vous êtes intéressé par l'étude des espèces et des fonctions des gènes, veuillez abandonner la recherche sur les algorithmes et utiliser directement les formules, ou utiliser directement les machines ;si vous êtes un hétéro en mathématiques et en ingénierie, n'hésitez pas.
méthode de la double courbe standard
Quantifiez le gène cible et le gène de ménage de l'échantillon témoin et de l'échantillon à tester à travers la courbe standard, puis calculez la valeur relative selon la formule de calcul, qui est le niveau d'expression relatif.
Avantages : analyse simple, optimisation expérimentale relativement simple
Inconvénient : Pour chaque gène, chaque série d'expériences doit faire une courbe standard
Application : L'une des deux méthodes quantitatives relatives les plus couramment utilisées et reconnues dans l'étude de la régulation de l'expression génique
La formule est la suivante :
Les exemples sont les suivants :
Calculer le montant relatif en fonction du résultat quantitatif
2 – Méthode △△Ct (méthode de comparaison des valeurs CT)
Avantages : Pas besoin de faire une courbe standard
Inconvénients : On suppose que le rendement d'amplification est proche de 100 % ;l'écart type est < 5 %, et la courbe standard et le rendement entre chaque amplification sont supposés cohérents ;l'optimisation des conditions expérimentales est plus compliquée.
Application : L'une des deux méthodes quantitatives relatives les plus couramment utilisées et reconnues dans l'étude de la régulation de l'expression génique
Bien sûr, l'efficacité d'amplification est généralement impossible à être parfaitement 1. Méthode de correction : Si nous savons que le gène cible et le gène de référence ont la même efficacité d'amplification, mais que l'efficacité d'amplification n'est pas égale à 1, alors 2-△△Ct peut être corrigé comme : (1+E )-△△Ct, par exemple, si l'efficacité d'amplification est de 0,95, alors la formule de calcul peut être corrigée à 1,95-△ △Ct
Jusqu'à présent, le contenu sur la PCR quantitative fluorescente est terminé .
Heure de publication : 06 avril 2023