Matériel de départ : ARN
La PCR de transcription inverse quantitative (RT-qPCR) est une méthode expérimentale utilisée dans les expériences de PCR utilisant l'ARN comme matériau de départ.Dans cette méthode, l'ARN total ou ARN messager (ARNm) est d'abord transcrit en ADN complémentaire (ADNc) par la transcriptase inverse.Par la suite, une réaction qPCR a été réalisée en utilisant l'ADNc comme matrice.La RT-qPCR a été utilisée dans diverses applications de biologie moléculaire, notamment l'analyse de l'expression génique, la validation des interférences ARN, la validation des microréseaux, la détection des agents pathogènes, les tests génétiques et la recherche sur les maladies.
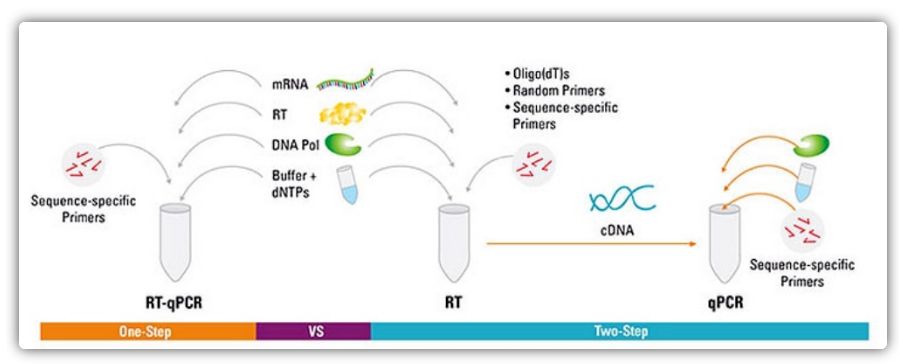
Méthodes en une et deux étapes pour la RT-qPCR
La RT-qPCR peut être réalisée par une méthode en une ou deux étapes.La RT-qPCR en une étape combine la transcription inverse et l'amplification par PCR, permettant à la transcriptase inverse et à l'ADN polymérase de terminer la réaction dans le même tube dans les mêmes conditions de tampon.La RT-qPCR en une étape ne nécessite que l'utilisation d'amorces spécifiques à la séquence.Dans la RT-qPCR en deux étapes, la transcription inverse et l'amplification par PCR sont effectuées dans deux tubes, en utilisant différents tampons optimisés, conditions de réaction et stratégies de conception d'amorces.
| Avantage | Désavantage | |
| Un pas | Cette méthode a moins d'erreur expérimentale car les deux réactions sont effectuées dans un seul tube
Moins d'étapes de pipetage réduisent le risque de contamination
Convient pour l'amplification/le criblage à haut débit, rapide et reproductible | Les réactions en deux étapes ne peuvent pas être optimisées séparément
Étant donné que les conditions de réaction sont compromises en combinant la réaction en deux étapes, la sensibilité n'est pas aussi bonne que celle de la méthode en deux étapes
Le nombre de cibles détectées par un seul échantillon est faible |
| Deux étapes | Capacité à créer des bibliothèques d'ADNc stables pouvant être stockées pendant de longues périodes et utilisées dans de multiples réactions
Les gènes cibles et les gènes de référence peuvent être amplifiés à partir de la même bibliothèque d'ADNc sans avoir besoin de plusieurs bibliothèques d'ADNc
Tampons de réaction et conditions de réaction qui permettent d'optimiser les cycles de réaction uniques
Sélection flexible des conditions de déclenchement | L'utilisation de plusieurs tubes et de plusieurs étapes de pipetage augmente le risque de contamination par l'ADN, et chronophage.
Nécessite plus d'optimisation que la méthode en une étape |
Produits connexes:
RT-qPCR Easyᵀᴹ (une étape)-SYBR Green I
RT-qPCR Easyᵀᴹ (une étape)-Taqman
RT Easyᵀᴹ I Master Premix pour la synthèse d'ADNC premier brin
Kit PCR en temps réel Easyᵀᴹ-SYBR Green I
PCR en temps réel Easyᵀᴹ-Taqman
Sélection de l'ARN total et de l'ARNm
Lors de la conception d'une expérience RT-qPCR, il est important de décider d'utiliser l'ARN total ou l'ARNm purifié comme modèle pour la transcription inverse.Bien que l'ARNm puisse être en mesure de fournir une sensibilité légèrement supérieure, l'ARN total est encore fréquemment utilisé.La raison en est que l'ARN total a un avantage plus important en tant que matériau de départ que l'ARNm.Premièrement, le processus nécessite moins d'étapes de purification, ce qui garantit une meilleure récupération quantitative de la matrice et une meilleure normalisation des résultats par rapport au nombre de cellules de départ.Deuxièmement, il évite l'étape d'enrichissement de l'ARNm, ce qui peut éviter la possibilité de résultats biaisés en raison de différentes récupérations de différents ARNm.Globalement, puisque dans la plupart des applications la quantification relative du gène cible est plus importante que la sensibilité absolue de la détection, l'ARN total est plus adapté dans la plupart des cas.
Amorce de transcription inverse
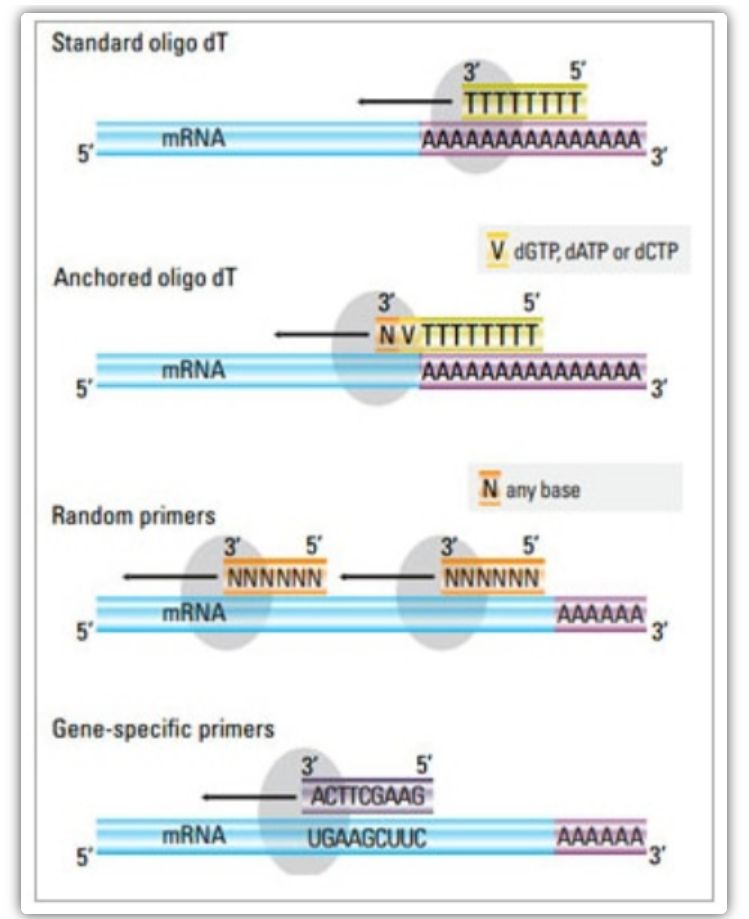
Dans la méthode en deux étapes, trois méthodes différentes peuvent être utilisées pour amorcer la réaction d'ADNc : les amorces oligo(dT), les amorces aléatoires ou les amorces spécifiques à la séquence.Typiquement, des amorces oligo(dT) et des amorces aléatoires sont utilisées en combinaison.Ces amorces s'hybrident au brin d'ARNm matrice et fournissent une transcriptase inverse avec un point de départ pour la synthèse.
| Sélection d'apprêt | La structure et la fonction | Avantage | Désavantage |
| Amorce oligo(dT) (ou amorce oligo(dT) ancrée) | Hybridation étendue aux résidus de thymine à la queue poly(A) de l'ARNm ;l'amorce oligo (dT) d'ancrage contient un G, C ou A à l'extrémité 3 '(site d'ancrage) | Synthèse d'ADNc pleine longueur à partir d'ARNm à queue poly(A)
Applicable lorsque moins de matière première est disponible
Le site d'ancrage garantit que l'amorce oligo (dT) se lie à la queue 5 'poly (A) de l'ARNm | Convient uniquement à l'amplification de gènes avec des queues poly(A)
Obtenir l'ADNc tronqué du site d'amorçage * 2 dans poly (A)
Biais pour se lier à l'extrémité 3′*
*Cette possibilité est minimisée si des amorces oligo(dT) ancrées sont utilisées |
| amorce aléatoire
| 6 à 9 bases de longueur, qui peuvent s'anneler à plusieurs sites pendant la transcription de l'ARN | Recuit à tous les ARN (ARNt, ARNr et ARNm)
Convient aux transcriptions avec une structure secondaire significative, ou lorsque moins de matériel de départ est disponible
Rendement élevé d'ADNc | L'ADNc est rétrotranscrit à partir de tous les ARN, ce qui n'est généralement pas souhaité et peut diluer le signal de l'ARNm cible
obtenir un ADNc tronqué |
| amorces spécifiques à la séquence | Amorces personnalisées ciblant des séquences d'ARNm spécifiques | bibliothèque d'ADNc spécifique
Améliorer la sensibilité
Utilisation d'amorces qPCR inverses | Uniquement limité à la synthèse d'un seul gène cible |
Transcriptase inverse
La transcriptase inverse est une enzyme qui utilise l'ARN pour synthétiser l'ADN.Certaines transcriptases inverses ont une activité RNase et peuvent dégrader les brins d'ARN dans les brins hybrides ARN-ADN après la transcription.S'il n'a pas d'activité enzymatique RNase, la RNaseH peut être ajoutée pour une efficacité qPCR plus élevée.Les enzymes couramment utilisées comprennent la transcriptase inverse du virus de la leucémie murine de Moloney et la transcriptase inverse du virus du myéloblastome aviaire.Pour la RT-qPCR, il est idéal de choisir une transcriptase inverse avec une thermostabilité plus élevée, de sorte que la synthèse d'ADNc puisse être effectuée à des températures plus élevées, assurant une transcription réussie des ARN avec une structure secondaire plus élevée, tout en maintenant leur pleine activité tout au long de la réaction, ce qui entraîne des rendements d'ADNc plus élevés.
Produits connexes:
Transcriptase inverse Foreasy M-MLV
Activité RNase H de la transcriptase inverse
La RNaseH est capable de dégrader les brins d'ARN des duplex ARN-ADN, permettant une synthèse efficace de l'ADN double brin.Cependant, lors de l'utilisation d'ARNm long comme matrice, l'ARN peut être dégradé prématurément, entraînant un ADNc tronqué.Par conséquent, il est souvent avantageux de minimiser l'activité RNaseH pendant le clonage d'ADNc si la synthèse de longs transcrits est souhaitée.En revanche, les transcriptases inverses à activité RNase H sont souvent bénéfiques pour les applications qPCR car elles améliorent la fusion des duplex ARN-ADN au cours du premier cycle de PCR.
Conception d'apprêt
Les amorces PCR utilisées pour l'étape qPCR dans la RT-qPCR devraient idéalement être conçues pour couvrir une jonction exon-exon, où une amorce d'amplification pourrait potentiellement couvrir une limite exon-intron réelle.Étant donné que les séquences d'ADN génomique contenant des introns ne sont pas amplifiées, cette conception réduit le risque de faux positifs amplifiés à partir de l'ADN génomique contaminant.
Si les amorces ne peuvent pas être conçues pour séparer les exons ou les limites exon-exon, il peut être nécessaire de traiter les échantillons d'ARN avec de la DNase I ou de la dsDNase sans RNase pour éliminer la contamination de l'ADN génomique.
Contrôle RT-qPCR
Un contrôle négatif de transcription inverse (contrôle -RT) doit être inclus dans toutes les expériences de RT-qPCR pour détecter la contamination par l'ADN (comme l'ADN génomique ou les produits de PCR des réactions précédentes).Ce contrôle contient tous les composants de la réaction à l'exception de la transcriptase inverse.Étant donné que la transcription inverse ne se produit pas avec ce contrôle, si une amplification par PCR est observée, la contamination par l'ADN est très probable.
Heure de publication : 02 août 2022








