- La PCR est une méthode utilisée pour amplifier l’ADN à partir d’une petite quantité de matrice d’ADN.La RT-PCR utilise la transcription inverse pour produire une matrice d'ADN à partir d'une source d'ARN qui peut ensuite être amplifiée.
- La PCR et la RT-PCR sont généralement des réactions à point final, tandis que la qPCR et la RT-qPCR utilisent la cinétique du taux de synthèse du produit au cours de la réaction PCR pour quantifier la quantité de matrice présente.
- Des méthodes plus récentes, telles que la PCR numérique, permettent une quantification absolue de la matrice d'ADN initiale, tandis que des méthodes telles que la PCR isotherme réduisent le besoin d'équipements coûteux pour fournir des résultats fiables.
La réaction en chaîne par polymérase (PCR) est une technique de biologie moléculaire relativement simple et largement utilisée pour amplifier et détecter des séquences d'ADN et d'ARN.Comparée aux méthodes traditionnelles de clonage et d’amplification de l’ADN, qui peuvent souvent prendre plusieurs jours, la PCR ne nécessite que quelques heures.La PCR est très sensible et nécessite un modèle minimal pour la détection et l'amplification de séquences spécifiques.Les méthodes de PCR de base ont progressé depuis la simple détection de l’ADN et de l’ARN.Ci-dessous, nous avons fourni un aperçu des différentes méthodes PCR et des réactifs que nous fournissons chez Enzo Life Sciences pour vos besoins de recherche.Notre objectif est d'aider les scientifiques à accéder rapidement aux réactifs PCR à utiliser dans leur prochain projet de recherche !
RAP
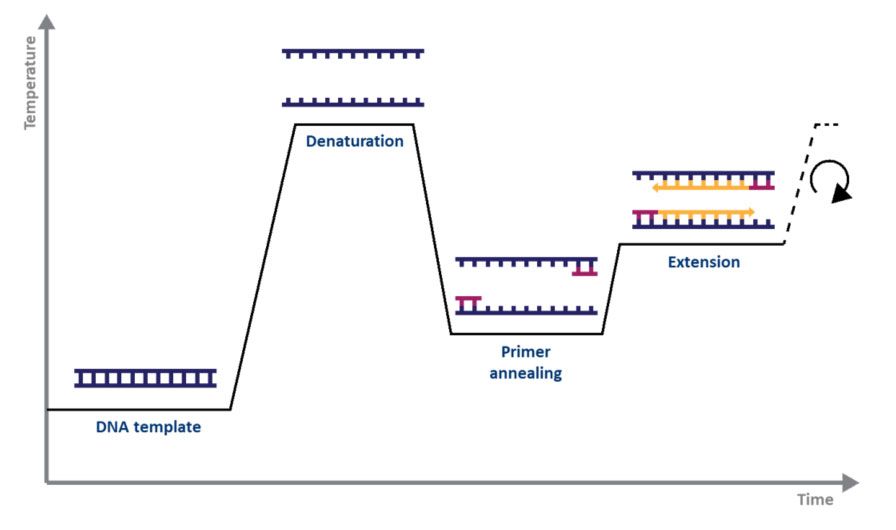
Pour la PCR standard, tout ce dont vous avez besoin est une ADN polymérase, du magnésium, des nucléotides, des amorces, la matrice d'ADN à amplifier et un thermocycleur.Le mécanisme de la PCR est aussi simple que son objectif : 1) l'ADN double brin (ADNdb) est dénaturé par la chaleur, 2) les amorces s'alignent sur les simples brins d'ADN et 3) les amorces sont étendues par l'ADN polymérase, ce qui donne deux copies de l'ADN polymérase. brin d'ADN d'origine.Le processus de dénaturation, de recuit et d’élongation sur une série de températures et de durées est connu sous le nom de cycle d’amplification (Fig. 1).
| Figure 1.Représentation schématique d'un cycle d'amplification par PCR. |
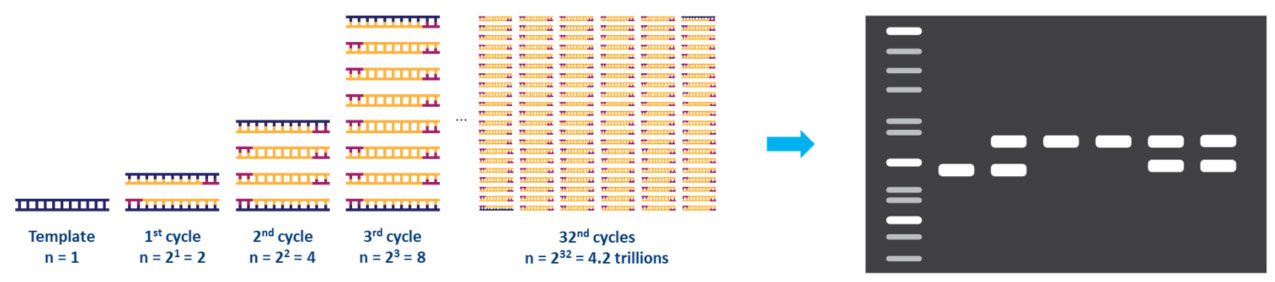
Chaque étape du cycle doit être optimisée pour le modèle et l’ensemble d’amorces utilisés.Ce cycle est répété environ 20 à 40 fois et le produit amplifié peut ensuite être analysé, généralement par gel d'agarose (Fig. 2).
| Figure 2.Amplification d'une matrice d'ADN par PCR et analyse par électrophorèse sur gel d'agarose. |
Étant donné que la PCR est une méthode très sensible et que de très petits volumes sont nécessaires pour des réactions uniques, la préparation d'un master mix pour plusieurs réactions est recommandée.Le mélange maître doit être bien mélangé, puis divisé en fonction du nombre de réactions, afin de garantir que chaque réaction contiendra la même quantité d'enzyme, de dNTP et d'amorces.De nombreux fournisseurs, comme Enzo Life Sciences, proposent également des mélanges PCR qui contiennent déjà tout sauf les amorces et la matrice d'ADN.
Les régions riches en guanine/cytosine (riches en GC) représentent un défi dans les techniques PCR standard.Les séquences riches en GC sont plus stables que les séquences avec une teneur plus faible en GC.De plus, les séquences riches en GC ont tendance à former des structures secondaires, telles que des boucles en épingle à cheveux.En conséquence, les doubles brins riches en GC sont difficiles à séparer complètement pendant la phase de dénaturation.Par conséquent, l’ADN polymérase ne peut pas synthétiser le nouveau brin sans entrave.Une température de dénaturation plus élevée peut améliorer cela, et des ajustements vers une température d'hybridation plus élevée et un temps d'hybridation plus court peuvent empêcher la liaison non spécifique des amorces riches en GC.Des réactifs supplémentaires peuvent améliorer l’amplification des séquences riches en GC.Le DMSO, le glycérol et la bétaïne aident à perturber les structures secondaires provoquées par les interactions GC et facilitent ainsi la séparation des doubles brins.
PCR à démarrage à chaud
L'amplification non spécifique est un problème qui peut survenir lors de la PCR.La plupart des ADN polymérases utilisées pour la PCR fonctionnent mieux à des températures comprises entre 68°C et 72°C.L'enzyme peut cependant également être active à des températures plus basses, bien qu'à un degré moindre.À des températures bien inférieures à la température d'hybridation, les amorces peuvent se lier de manière non spécifique et conduire à une amplification non spécifique, même si la réaction se déroule sur de la glace.Cela peut être évité en utilisant des inhibiteurs de polymérase qui se dissocient de l'ADN polymérase seulement une fois qu'une certaine température est atteinte, d'où le terme PCR à démarrage à chaud.L'inhibiteur peut être un anticorps qui se lie à la polymérase et se dénature à la température de dénaturation initiale (95 °C généralement).
Polymérase haute fidélité
Bien que les ADN polymérases s'amplifient assez précisément par rapport à la séquence matrice d'origine, des erreurs dans l'appariement des nucléotides peuvent survenir.Des inadéquations dans des applications telles que le clonage peuvent entraîner des transcriptions tronquées et des protéines mal traduites ou inactives en aval.Pour éviter ces décalages, des polymérases ayant une activité de « relecture » ont été identifiées et intégrées au workflow.La première polymérase de relecture, Pfu, a été identifiée en 1991 chez Pyrococcus furiosus.Cette enzyme Pfu a une activité exonucléase 3' à 5'.Au fur et à mesure que l'ADN est amplifié, l'exonucléase élimine les nucléotides incompatibles à l'extrémité 3' du brin.Le bon nucléotide est ensuite remplacé et la synthèse de l'ADN se poursuit.L'identification de séquences nucléotidiques incorrectes repose sur l'affinité de liaison du nucléoside triphosphate correct avec l'enzyme, où une liaison inefficace ralentit la synthèse et permet le remplacement correct.L'activité de relecture de la polymérase Pfu entraîne moins d'erreurs dans la séquence finale par rapport à l'ADN polymérase Taq.Ces dernières années, d'autres enzymes de relecture ont été identifiées et des modifications de l'enzyme Pfu originale ont été apportées pour réduire davantage le taux d'erreur lors de l'amplification de l'ADN.
RT-PCR
La PCR par transcription inverse, ou RT-PCR, permet d'utiliser l'ARN comme matrice.Une étape supplémentaire permet la détection et l’amplification de l’ARN.L'ARN est transcrit de manière inverse en ADN complémentaire (ADNc), à l'aide de la transcriptase inverse.La qualité et la pureté de la matrice d’ARN sont essentielles au succès de la RT-PCR.La première étape de la RT-PCR est la synthèse d’un hybride ADN/ARN.La transcriptase inverse possède également une fonction RNase H, qui dégrade la partie ARN de l'hybride.La molécule d'ADN simple brin est ensuite complétée par l'activité ADN polymérase ADN-dépendante de la transcriptase inverse en ADNc.L'efficacité de la réaction du premier brin peut affecter le processus d'amplification.À partir de là, la procédure PCR standard est utilisée pour amplifier l’ADNc.La possibilité de transformer l’ARN en ADNc par RT-PCR présente de nombreux avantages et est principalement utilisée pour l’analyse de l’expression génique.L’ARN est simple brin et très instable, ce qui rend son travail difficile.Il sert généralement de première étape dans la qPCR, qui quantifie les transcrits d’ARN dans un échantillon biologique.
qPCR et RT-qPCR
La PCR quantitative (qPCR) est utilisée pour détecter, caractériser et quantifier les acides nucléiques pour de nombreuses applications.Dans la RT-qPCR, les transcrits d’ARN sont souvent quantifiés en les transcrivant d’abord en ADNc, comme décrit ci-dessus, puis en effectuant une qPCR.Comme dans la PCR standard, l'ADN est amplifié par trois étapes répétitives : dénaturation, recuit et allongement.Cependant, dans la qPCR, le marquage fluorescent permet la collecte de données au fur et à mesure de la progression de la PCR.Cette technique présente de nombreux avantages en raison de la gamme de méthodes et de produits chimiques disponibles.
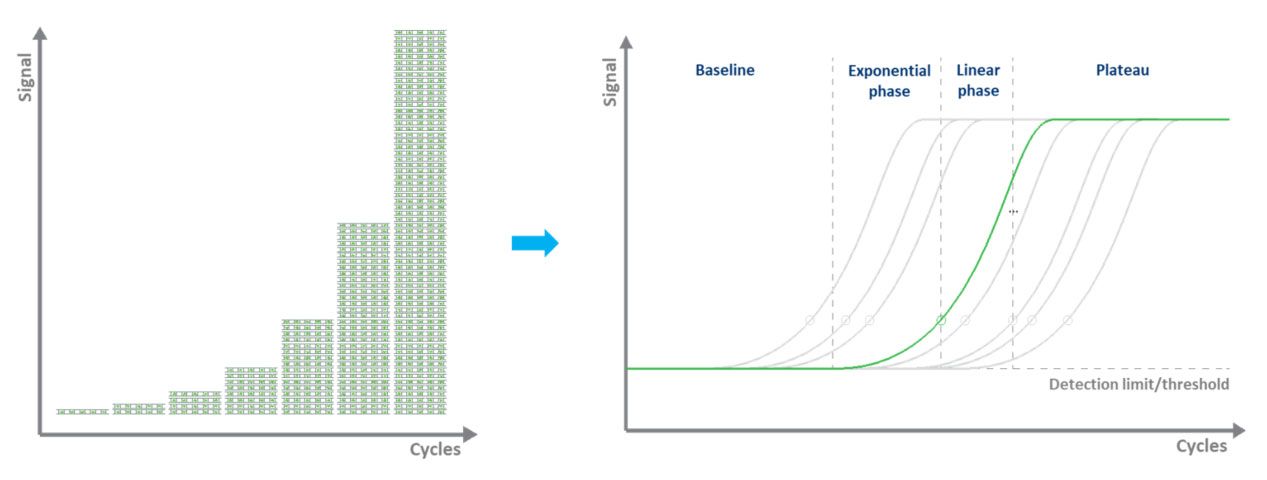
Dans la qPCR à base de colorant (généralement vert), le marquage fluorescent permet la quantification des molécules d'ADN amplifiées en utilisant un colorant se liant à l'ADNdb.Lors de chaque cycle, la fluorescence est mesurée.Le signal de fluorescence augmente proportionnellement à la quantité d'ADN répliqué.Ainsi, l’ADN est quantifié « en temps réel » (Fig. 3).Les inconvénients de la qPCR à base de colorant sont qu’une seule cible peut être examinée à la fois et que le colorant se liera à tout ADN double- brin présent dans l’échantillon.
| Figure 3.Amplifier une matrice d'ADN par qPCR et mesurer le signal de fluorescence en temps réel. |
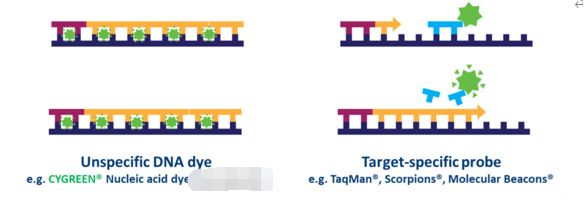
Dans la qPCR basée sur des sondes, de nombreuses cibles peuvent être détectées simultanément dans chaque échantillon, mais cela nécessite l'optimisation et la conception d'une ou plusieurs sondes spécifiques à la cible utilisées en plus des amorces.Plusieurs types de sondes sont disponibles, mais le type le plus courant est une sonde d'hydrolyse, qui intègre un fluorophore et un extincteur.Le transfert d'énergie par résonance de fluorescence (FRET) empêche l'émission du fluorophore via le quencher lorsque la sonde est intacte.Cependant, lors de la réaction PCR, la sonde est hydrolysée lors de l'extension de l'amorce et de l'amplification de la séquence spécifique à laquelle elle est liée.Le clivage de la sonde sépare le fluorophore du quencher et entraîne une augmentation de la fluorescence dépendante de l'amplification (Fig. 4).Ainsi, le signal de fluorescence provenant d’une réaction qPCR basée sur une sonde est proportionnel à la quantité de séquence cible de la sonde présente dans l’échantillon.Étant donné que la qPCR basée sur une sonde est plus spécifique que la qPCR basée sur un colorant, c'est souvent la technologie utilisée dans les tests de diagnostic basés sur la qPCR.
| Graphique 4.Différences entre la qPCR à base de colorant et la qPCR à base de sonde. |
Amplification isotherme
Les techniques de PCR mentionnées ci-dessus nécessitent un équipement de thermocyclage coûteux pour augmenter et diminuer avec précision les températures de la chambre pour les étapes de dénaturation, de recuit et d'extension.Un certain nombre de techniques ont été développées qui ne nécessitent pas de dispositifs aussi précis et peuvent être réalisées dans un simple bain-marie ou même dans les cellules d'intérêt.Ces techniques sont collectivement appelées amplification isotherme et fonctionnent sur la base d’une amplification exponentielle, linéaire ou en cascade.
Le type d’amplification isotherme le plus connu est l’amplification isotherme à médiation par boucle, ou LAMP.LAMP utilise une amplification exponentielle à 65⁰C pour amplifier l’ADN ou l’ARN matrice.Lors de l'exécution de LAMP, quatre à six amorces complémentaires aux régions de l'ADN cible sont utilisées avec une ADN polymérase pour synthétiser un nouvel ADN.Deux de ces amorces ont des séquences complémentaires qui reconnaissent les séquences des autres amorces et les lient, permettant ainsi la formation d’une structure en « boucle » dans l’ADN nouvellement synthétisé qui facilite ensuite l’hybridation des amorces lors des cycles d’amplification ultérieurs.LAMP peut être visualisé par plusieurs méthodes, notamment la fluorescence, l’électrophorèse sur gel d’agarose ou la colorimétrie.La facilité de visualisation et de détection de la présence ou de l'absence du produit par colorimétrie et l'absence d'équipement coûteux requis ont fait de LAMP une option appropriée pour les tests du SRAS-CoV-2 dans les zones où les tests cliniques en laboratoire n'étaient pas facilement disponibles, ou pour le stockage et le transport des échantillons. n'était pas réalisable, ou dans des laboratoires qui ne disposaient pas auparavant d'équipement de thermocyclage PCR.
Heure de publication : 19 août 2023