1. Connaissances de base (si vous souhaitez voir la partie expérimentale, merci de passer directement à la deuxième partie)
En tant que réaction dérivée de la PCR conventionnelle, la PCR en temps réel surveille principalement le changement de la quantité de produit d'amplification dans chaque cycle de la réaction d'amplification PCR en temps réel grâce au changement du signal de fluorescence et analyse quantitativement le modèle de départ grâce à la relation entre la valeur ct et la courbe standard .
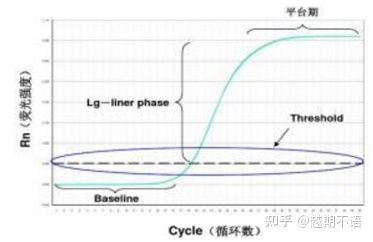
Les données spécifiques de la RT-PCR sontligne de base, seuil de fluorescenceetValeur CT.
| ligne de base : | La valeur de fluorescence du 3e au 15e cycle est la ligne de base (ligne de base), qui est causée par l'erreur occasionnelle de la mesure. |
| Seuil (seuil): | Fait référence à la limite de détection de fluorescence fixée à une position appropriée dans la région de croissance exponentielle de la courbe d'amplification, généralement 10 fois l'écart type de la ligne de base. |
| Valeur CT : | C'est le nombre de cycles de PCR lorsque la valeur de fluorescence dans chaque tube de réaction atteint le seuil. La valeur Ct est inversement proportionnelle à la quantité de modèle initial. |
Méthodes de marquage courantes pour la RT-PCR :
| méthode | avantage | défaut | champ d'application |
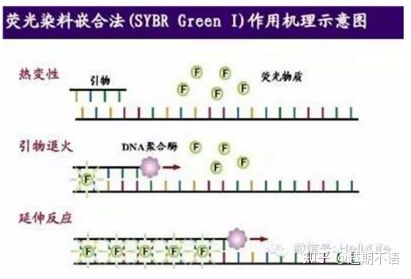
| SYBR VertⅠ | Large applicabilité, sensible, bon marché et pratique | Les exigences en matière d'apprêt sont élevées, sujettes à des bandes non spécifiques | Il convient à l'analyse quantitative de divers gènes cibles, à la recherche sur l'expression des gènes et à la recherche sur les animaux et les plantes recombinants transgéniques. |
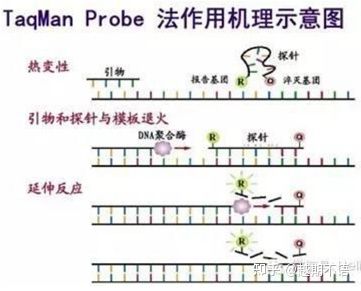
| TaqMan | Bonne spécificité et haute répétabilité | Le prix est élevé et ne convient qu'à des objectifs spécifiques. | Détection d'agents pathogènes, recherche sur les gènes de résistance aux médicaments, évaluation de l'efficacité des médicaments, diagnostic des maladies génétiques. |
| balise moléculaire | Haute spécificité, fluorescence, faible bruit de fond | Le prix est élevé, il ne convient qu'à un usage spécifique, la conception est difficile et le prix est élevé. | Analyse de gènes spécifiques, analyse SNP |
2. Étapes expérimentales
2.1 À propos du regroupement expérimental- il doit y avoir plusieurs puits dans le groupe, et il doit y avoir des répétitions biologiques.
| ① | Contrôle vide | Utilisé pour détecter l'état de croissance cellulaire dans les expériences |
| ② | siRNA contrôle négatif (séquence siRNA non spécifique) | Démontrer la spécificité de l'action de l'ARNi.L'ARNsi peut induire une réponse au stress non spécifique à une concentration de 200 nM. |
| ③ | Contrôle des réactifs de transfection | Exclure la toxicité du réactif de transfection pour les cellules ou l'effet sur l'expression du gène cible |
| ④ | siRNA contre le gène cible | Abattre l'expression du gène cible |
| ⑤ (facultatif) | ARNsi positif | Utilisé pour dépanner le système expérimental et les problèmes opérationnels |
| ⑥ (facultatif) | ARNsi de contrôle fluorescent | L'efficacité de la transfection cellulaire peut être observée au microscope |
2.2 Principes de conception des amorces
| Taille de fragment amplifié | De préférence à 100-150bp |
| Longueur d'amorce | 18-25pb |
| Contenu GC | 30%-70%, de préférence 45%-55% |
| Valeur Tm | 58-60℃ |
| Séquence | Éviter T/C continu ;A/G continu |
| 3 fin de séquence | Évitez les riches en GC ou en AT ;la base terminale est de préférence G ou C ;il vaut mieux éviter T |
| Complémentarité | Éviter les séquences complémentaires de plus de 3 bases dans l'amorce ou entre deux amorces |
| Spécificité | Utiliser la recherche par explosion pour confirmer la spécificité de l'amorce |
①SiRNA est spécifique à l'espèce et les séquences des différentes espèces seront différentes.
②SiRNA est emballé dans une poudre lyophilisée, qui peut être stockée de manière stable pendant 2 à 4 semaines à température ambiante.
2.3 Outils ou réactifs qui doivent être préparés à l'avance
| Abécédaire (référence interne) | Y compris avant et arrière deux |
| Amorces (gène cible) | Y compris avant et arrière deux |
| Cible Si ARN (3 barrettes) | Généralement, l'entreprise va synthétiser 3 barrettes, puis choisir l'une des trois par RT-PCR |
| Trousse de transfection | Lipo2000 etc... |
| Kit d'extraction rapide d'ARN | Pour l'extraction d'ARN après transfection |
| Kit de transcription inverse rapide | pour la synthèse d'ADNc |
| Kit d'amplification PCR | 2 × Super SYBR Vert Mélange maître qPCR |
2.4 En ce qui concerne les questions auxquelles il faut prêter attention dans les étapes expérimentales spécifiques :
①Processus de transfection d'ARNsi
1. Pour le placage, vous pouvez choisir une plaque à 24 puits, une plaque à 12 puits ou une plaque à 6 puits (la concentration moyenne d'ARN proposée dans chaque puits d'une plaque à 24 puits est d'environ 100 à 300 ng/uL), et la densité de transfection optimale des cellules est de 60 % à 80 % environ.
2. Les étapes de transfection et les exigences spécifiques sont strictement conformes aux instructions.
3. Après la transfection, les échantillons peuvent être prélevés dans les 24 à 72 heures pour la détection de l'ARNm (RT-PCR) ou la détection des protéines dans les 48 à 96 heures (WB)
② Processus d'extraction d'ARN
1. Empêcher la contamination par des enzymes exogènes.Cela comprend principalement le port strict de masques et de gants ;utiliser des embouts de pipette et des tubes EP stérilisés ;l'eau utilisée dans l'expérience doit être exempte de RNase.
2. Il est recommandé de faire deux fois ce qui est suggéré dans le kit d'extraction rapide, ce qui améliorera vraiment la pureté et le rendement.
3. Les déchets liquides ne doivent pas toucher la colonne d'ARN.
③ Quantification de l'ARN
Une fois l'ARN extrait, il peut être quantifié directement avec Nanodrop et la lecture minimale peut être aussi faible que 10 ng/ul.
④Processus de transcription inverse
1. En raison de la sensibilité élevée de la RT-qPCR, au moins 3 puits parallèles doivent être créés pour chaque échantillon afin d'éviter que le Ct ultérieur soit trop différent ou que l'écart-type soit trop grand pour une analyse statistique.
2. Ne pas congeler et décongeler le mélange maître à plusieurs reprises.
3. Chaque tube/trou doit être remplacé par un nouvel embout !N'utilisez pas continuellement la même pointe de pipette pour ajouter des échantillons !
4. Le film attaché à la plaque 96 puits après l'ajout de l'échantillon doit être lissé avec une plaque.Il est préférable de centrifuger avant de le mettre sur la machine, afin que le liquide sur la paroi du tube puisse s'écouler et éliminer les bulles d'air.
⑤Analyse de courbe commune
| Pas de période de croissance logarithmique | Concentration éventuellement élevée de gabarit |
| Pas de valeur CT | Étapes incorrectes pour détecter les signaux fluorescents ; dégradation des amorces ou des sondes - son intégrité peut être détectée par électrophorèse PAGE ; quantité insuffisante de modèle ; dégradation des matrices - évitant l'introduction d'impuretés et la congélation et la décongélation répétées lors de la préparation des échantillons ; |
| CT>38 | Faible efficacité d'amplification ;Le produit PCR est trop long ;divers composants de la réaction sont dégradés |
| Courbe d'amplification linéaire | Les sondes peuvent être partiellement dégradées par des cycles répétés de congélation-décongélation ou une exposition prolongée à la lumière |
| La différence de trous en double est particulièrement grande | La solution réactionnelle n'est pas complètement fondue ou la solution réactionnelle n'est pas mélangée ;le bain thermal de l'instrument PCR est contaminé par des substances fluorescentes |
2.5 À propos de l'analyse des données
L'analyse des données de qPCR peut être divisée en quantification relative et quantification absolue.Par exemple, les cellules du groupe de traitement par rapport aux cellules du groupe témoin,
Combien de fois l'ARNm du gène X change, c'est une quantification relative ;dans un certain nombre de cellules, l'ARNm du gène X
Combien d'exemplaires il y a, c'est une quantification absolue.Habituellement, ce que nous utilisons le plus au laboratoire est la méthode quantitative relative.Généralement,la méthode 2-ΔΔctest le plus utilisé dans les expériences , seule cette méthode sera donc présentée en détail ici.
Méthode 2-ΔΔct : Le résultat obtenu est la différence d'expression du gène cible dans le groupe expérimental par rapport au gène cible dans le groupe témoin.Il est nécessaire que les efficacités d'amplification du gène cible et du gène de référence interne soient proches de 100 %, et l'écart relatif ne doit pas dépasser 5 %.
La méthode de calcul est la suivante :
Δct groupe témoin = valeur ct du gène cible dans le groupe témoin – valeur ct du gène de référence interne dans le groupe témoin
Δct groupe expérimental = valeur ct du gène cible dans le groupe expérimental – valeur ct du gène de référence interne dans le groupe expérimental
ΔΔct=Δct groupe expérimental-Δct groupe témoin
Enfin, calculez le multiple de la différence de niveau d'expression :
Change Fold=2-ΔΔct (correspondant à la fonction excel est POWER)
Produits connexes:
Heure de publication : 20 mai 2023