Après que le chercheur américain Eric S. Lander a officiellement proposé le polymorphisme mononucléotidique (SNP) comme marqueur moléculaire de troisième génération en 1996, le SNP a été largement utilisé dans l'analyse d'association de traits économiques, la construction de cartes de liaison génétique biologique et le dépistage des gènes pathogènes humains., diagnostic et prédiction du risque de maladie, dépistage individualisé des médicaments et autres domaines de recherche biologique et médicale.Dans le domaine de la sélection des cultures de rente, la détection de SNP peut réaliser une sélection précoce des caractères requis.Cette sélection a les caractéristiques d'une grande précision et peut efficacement éviter l'interférence de la morphologie et des facteurs environnementaux, raccourcissant ainsi considérablement le processus de sélection.Par conséquent, le SNP joue un rôle énorme dans le domaine de la recherche fondamentale.
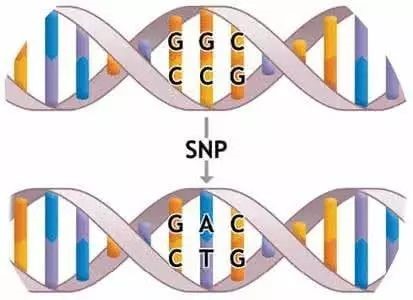
Le polymorphisme de nucléotide unique (polymorphisme de nucléotide unique, SNP) fait référence au phénomène selon lequel il existe des différences de nucléotide unique dans la même position dans la séquence d'ADN d'individus de la même espèce ou d'espèces différentes.L'insertion, la suppression, la conversion et l'inversion d'une seule base peuvent toutes provoquer cette différence.Dans le passé, la définition du SNP était différente de celle de la mutation.Un locus variant nécessite que la fréquence d'un des allèles dans la population soit supérieure à 1% pour être défini comme un locus SNP.Cependant, avec l'expansion des théories biologiques modernes et l'application de la technologie, la fréquence des allèles n'est plus une condition nécessaire pour limiter la définition du SNP.Selon les données de variation d'un seul nucléotide incluses dans la base de données Single Nucleotide Polymorphisms (dbSNP) du National Center for Biotechnology Information (NCBI), l'insertion/suppression à basse fréquence, la variation des microsatellites, etc. sont également incluses.
Dans le corps humain, la fréquence des SNP est de 0,1 %.En d'autres termes, il y a en moyenne un site SNP pour 1000 paires de bases.Bien que la fréquence d'occurrence soit relativement élevée, tous les sites SNP ne peuvent pas être des marqueurs candidats liés aux traits.Ceci est principalement lié à l'emplacement où le SNP se produit.
Théoriquement, les SNP peuvent apparaître n'importe où dans la séquence du génome.Les SNP présents dans la région codante peuvent produire des mutations synonymes et des mutations non synonymes, c'est-à-dire que l'acide aminé change ou non avant et après la mutation.L'acide aminé modifié fait généralement perdre à la chaîne peptidique sa fonction d'origine (mutation faux-sens) et peut également provoquer un arrêt de la traduction (mutation non-sens).Les SNP qui se produisent dans les régions non codantes et les régions intergéniques peuvent affecter l'épissage de l'ARNm, la composition de la séquence d'ARN non codante et l'efficacité de liaison des facteurs de transcription et de l'ADN.La relation spécifique est illustrée dans la figure :
Types de SNP :
Plusieurs méthodes courantes de typage SNP et leur comparaison
Selon différents principes, les méthodes courantes de détection de SNP sont divisées dans les catégories suivantes :
Comparaison de classification des méthodes de détection
Remarque : Les méthodes de détection de SNP les plus courantes sont actuellement utilisées, d'autres méthodes de détection telles que l'hybridation de site spécifique (ASH), l'extension d'amorce de site spécifique (ASPE), l'extension de base unique (SBCE), la coupe de site spécifique (ASC), la technologie des puces génétiques, la technologie de spectrométrie de masse, etc. n'ont pas été classées ni comparées.
Le coût et le temps de purification des acides nucléiques dans les plusieurs méthodes de détection de SNP courantes ci-dessus sont inévitables.Cependant, les kits associés basés sur la technologie PCR directe de Foregene peuvent effectuer directement une amplification PCR ou qPCR sur des échantillons non purifiés, ce qui apporte une commodité sans précédent à la détection des SNP.
Les produits de la série PCR directe de Foregene omettent simplement et grossièrement les étapes de purification des échantillons, ce qui réduit considérablement le temps et le coût nécessaires à la préparation des modèles.La polymérase Taq unique a une excellente capacité d'amplification et peut tolérer une variété d'inhibiteurs provenant d'environnements d'amplification complexes.Ces caractéristiques offrent une garantie technique pour l'obtention de produits spécifiques à haut rendement.Kits Foregene Direct PCR/qPCR pour différents types d'échantillons, tels que : tissus animaux (queue de rat, poisson zèbre, etc.), feuilles de plantes, graines (dont échantillons de polysaccharides et de polyphénols), etc.
Heure de publication : 23 juillet 2021