一、Augmenter la sensibilité du système de réaction:
1. Isoler l'ARN de haute qualité:
La synthèse réussie d'ADNc provient d'un ARN de haute qualité.L'ARN de haute qualité doit au moins être complet et exempt d'inhibiteurs de la transcriptase inverse tels que l'EDTA ou le SDS.La qualité de l'ARN détermine la quantité maximale d'informations de séquence que vous pouvez transcrire en ADNc.Une méthode courante de purification d'ARN est une méthode en une étape utilisant de l'isothiocyanate de guanidine/phénol acide.Pour éviter la contamination par des traces de RNase, l'ARN isolé à partir d'échantillons riches en RNase (tels que le pancréas) doit être stocké dans du formaldéhyde pour préserver un ARN de haute qualité, en particulier pour un stockage à long terme.L'ARN extrait du foie de rat a été fondamentalement dégradé après avoir été stocké dans l'eau pendant une semaine, tandis que l'ARN extrait de la rate de rat est resté stable après avoir été stocké dans l'eau pendant 3 ans.De plus, les transcrits de plus de 4 kb sont plus sensibles à la dégradation par les traces de RNases que les petits transcrits.Pour augmenter la stabilité des échantillons d'ARN stockés, l'ARN peut être dissous dans du formamide déionisé et stocké à -70°C.Le formamide utilisé pour conserver l'ARN doit être exempt de débris dégradant l'ARN.L'ARN du pancréas peut être conservé dans du formamide pendant au moins un an.Lorsque vous vous préparez à utiliser l'ARN, vous pouvez utiliser la méthode suivante pour précipiter l'ARN : ajouter du NaCl à 0,2 M et 4 fois le volume d'éthanol, placer à température ambiante pendant 3 à 5 minutes et centrifuger à 10 000 x g pendant 5 minutes.
2. Utilisez la transcriptase inverse RNaseH-inactive (RNaseH-):
Des inhibiteurs de RNase sont souvent ajoutés aux réactions de transcription inverse pour augmenter la longueur et le rendement de la synthèse d'ADNc.Les inhibiteurs de la RNase doivent être ajoutés lors de la réaction de synthèse du premier brin en présence d'un tampon et d'un agent réducteur (tel que le DTT), car le processus précédant la synthèse de l'ADNc dénature l'inhibiteur, libérant ainsi la RNase liée qui peut dégrader l'ARN.Les inhibiteurs de la protéine RNase empêchent uniquement la dégradation de l'ARN par la RNase A, B, C, et n'empêchent pas la RNase sur la peau, alors faites attention à ne pas introduire de RNase de vos doigts malgré l'utilisation de ces inhibiteurs.
La transcriptase inverse catalyse la conversion de l'ARN en ADNc.M-MLV et AMV ont tous deux une activité RNaseH endogène en plus de leur propre activité polymérase.L'activité RNaseH et l'activité polymérase se font concurrence pour le brin hybride formé entre la matrice d'ARN et l'amorce d'ADN ou le brin d'extension d'ADNc, et dégradent le brin d'ARN dans le complexe ARN: ADN.La matrice d'ARN dégradée par l'activité RNaseH ne peut plus servir de substrat efficace pour la synthèse d'ADNc, ce qui réduit le rendement et la durée de la synthèse d'ADNc.Par conséquent, il serait avantageux d'éliminer ou de réduire considérablement l'activité RNaseH de la transcriptase inverse.。
La transcriptase inverse SuperScript Ⅱ, la transcriptase inverse RNaseH-MMLV et la transcriptase inverse thermoScript, RNaseH-AMV, peuvent obtenir une plus grande quantité et plus d'ADNc complet que le MMLV et l'AMV.La sensibilité de la RT-PCR sera affectée par la quantité de synthèse d'ADNc.ThermoScript est beaucoup plus sensible qu'AMV.La taille des produits de RT-PCR est limitée par la capacité de la transcriptase inverse à synthétiser l'ADNc, en particulier lors du clonage d'ADNc plus grands.Par rapport au MMLV, SuperScripⅡ a considérablement augmenté le rendement des produits de RT-PCR longs.La transcriptase inverse RNaseH a également une thermostabilité accrue, de sorte que la réaction peut être effectuée à des températures supérieures aux 37-42°C normaux.Dans les conditions de synthèse suggérées, utilisez une amorce oligo(dT) et 10 μCi de [α-P]dCTP.Le rendement total du premier brin a été calculé en utilisant la méthode de précipitation au TCA.L'ADNc de pleine longueur a été analysé en utilisant des bandes triées par taille excisées et comptées sur un gel d'agarose alcalin.
3. Augmentez la température d'incubation pour la transcription inverse:
Une température d'incubation plus élevée aide à ouvrir la structure secondaire de l'ARN, augmentant le rendement de la réaction.Pour la plupart des matrices d'ARN, l'incubation de l'ARN et des amorces à 65 °C sans tampon ni sel, suivie d'un refroidissement rapide sur de la glace éliminera la plupart des structures secondaires et permettra aux amorces de se lier.Cependant, certains modèles ont encore des structures secondaires, même après dénaturation par la chaleur.L'amplification de ces modèles difficiles peut être effectuée à l'aide de la transcriptase inverse ThermoScript et en plaçant la réaction de transcription inverse à une température plus élevée pour améliorer l'amplification.Des températures d'incubation plus élevées peuvent également augmenter la spécificité, en particulier lorsque des amorces spécifiques à un gène (GSP) sont utilisées pour la synthèse d'ADNc (voir chapitre 3).Si vous utilisez GSP, assurez-vous que la Tm des amorces est la même que la température d'incubation attendue.Ne pas utiliser d'amorces oligo(dT) et aléatoires au-dessus de 60°C.Les amorces aléatoires nécessitent une incubation à 25°C pendant 10 minutes avant d'augmenter à 60°C.En plus d'utiliser une température de transcription inverse plus élevée, la spécificité peut également être améliorée en transférant directement le mélange ARN/amorce de la température de dénaturation de 65 ° C à la température d'incubation de transcription inverse et en ajoutant un mélange réactionnel préchauffé 2 × (synthèse de démarrage à chaud d'ADNc) .Cette approche permet d'éviter l'appariement de bases intermoléculaires qui se produit à des températures plus basses.La commutation de température multiple requise pour la RT-PCR peut être simplifiée en utilisant un thermocycleur.
La Tth polymérase thermostable agit comme une ADN polymérase en présence de Mg2+ et comme une ARN polymérase en présence de Mn2+.Il peut être maintenu au chaud à une température maximale de 65°C.Cependant, la présence de Mn2+ pendant la PCR réduit la fidélité, ce qui rend la polymérase Tth moins adaptée à une amplification de haute précision, telle que le clonage d'ADNc.De plus, Tth a une faible efficacité de transcription inverse, ce qui réduit la sensibilité, et, puisque la transcription inverse et la PCR peuvent être réalisées avec une seule enzyme, les réactions de contrôle sans transcription inverse ne peuvent pas être utilisées pour comparer les produits d'amplification d'ADNc avec l'ADN génomique contaminant.Les produits d'amplification ont été séparés.
4. Additifs qui favorisent la transcription inverse:
Des additifs comprenant du glycérol et du DMSO sont ajoutés à la réaction de synthèse du premier brin, ce qui peut réduire la stabilité du double brin d'acide nucléique et délier la structure secondaire de l'ARN.Jusqu'à 20 % de glycérol ou 10 % de DMSO peuvent être ajoutés sans affecter l'activité SuperScript II ou MMLV.L'AMV peut également tolérer jusqu'à 20% de glycérol sans perte d'activité.Afin de maximiser la sensibilité de la RT-PCR dans la réaction de transcription inverse SuperScriptⅡ, 10 % de glycérol peuvent être ajoutés et incubés à 45 °C.Si 1/10 du produit de la réaction de transcription inverse est ajouté à la PCR, alors la concentration de glycérol dans la réaction d'amplification est de 0,4 %, ce qui n'est pas suffisant pour inhiber la PCR.
5. Traitement RNaseH:
Le traitement des réactions de synthèse d'ADNc avec la RNaseH avant la PCR peut augmenter la sensibilité.Pour certaines matrices, on pense que l'ARN dans la réaction de synthèse d'ADNc empêche la liaison des produits d'amplification, auquel cas le traitement à la RNaseH peut augmenter la sensibilité.En règle générale, un traitement à la RNaseH est nécessaire lors de l'amplification de matrices cibles d'ADNc pleine longueur plus longues, telles que la schérose tubéreuse II à faible nombre de copies.Pour cette matrice difficile, le traitement à la RNaseH a amélioré le signal produit par SuperScript II ou l'ADNc synthétisé par AMV.Pour la plupart des réactions RT-PCR, le traitement à la RNaseH est facultatif, car l'étape de dénaturation par PCR à 95°C hydrolyse généralement l'ARN dans le complexe ARN : ADN.
6. Amélioration de la méthode de détection des petits ARN:
La RT-PCR est particulièrement difficile lorsque seules de petites quantités d'ARN sont disponibles.Le glycogène ajouté comme support lors de l'isolement de l'ARN contribue à augmenter le rendement des petits échantillons.Du glycogène sans RNase peut être ajouté en même temps que Trizol.Le glycogène est soluble dans l'eau et peut être conservé dans la phase aqueuse avec de l'ARN pour faciliter la précipitation ultérieure.Pour les échantillons de moins de 50 mg de tissu ou 106 cellules cultivées, la concentration recommandée de glycogène sans RNase est de 250 μg/ml.
L'ajout de BSA acétylé à la réaction de transcription inverse à l'aide de SuperScript II peut augmenter la sensibilité, et pour de petites quantités d'ARN, la réduction de la quantité de SuperScript II et l'ajout de 40 unités d'inhibiteur de nucléase RNaseOut peuvent augmenter le niveau de détection.Si du glycogène est utilisé dans le processus d'isolement de l'ARN, il est toujours recommandé d'ajouter de la BSA ou un inhibiteur de RNase lors de l'utilisation de SuperScript II pour la réaction de transcription inverse.
二、Augmenter la spécificité de la RT-PCR
1. Asynthèse CND:
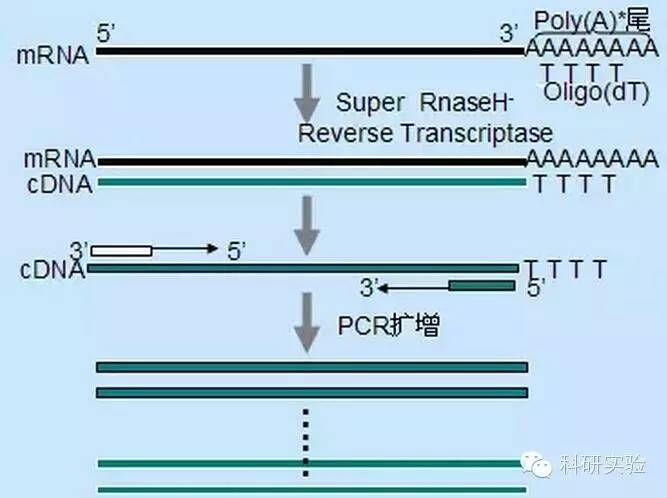
La synthèse d'ADNc du premier brin peut être initiée en utilisant trois méthodes différentes, dont la spécificité relative affecte la quantité et le type d'ADNc synthétisé.
La méthode des amorces aléatoires était la moins spécifique des trois méthodes.Les amorces s'hybrident sur plusieurs sites tout au long du transcrit, générant des ADNc courts et de longueur partielle.Cette méthode est fréquemment utilisée pour obtenir des séquences d'extrémité 5' et pour obtenir des ADNc à partir de matrices d'ARN avec des régions de structure secondaire ou avec des sites de terminaison qui ne peuvent pas être répliqués par la transcriptase inverse.Pour obtenir l'ADNc le plus long, le rapport des amorces à l'ARN dans chaque échantillon d'ARN doit être déterminé de manière empirique.La concentration de départ des amorces aléatoires variait de 50 à 250 ng par réaction de 20 μl.Etant donné que l'ADNc synthétisé à partir d'ARN total en utilisant des amorces aléatoires est principalement de l'ARN ribosomal, l'ARN poly(A)+ est généralement choisi comme matrice.
Les amorces oligo(dT) sont plus spécifiques que les amorces aléatoires.Il s'hybride à la queue poly (A) trouvée à l'extrémité 3 'de la plupart des ARNm eucaryotes.Étant donné que l'ARN poly(A)+ représente environ 1 % à 2 % de l'ARN total, la quantité et la complexité de l'ADNc sont bien moindres qu'avec des amorces aléatoires.En raison de sa grande spécificité, l'oligo(dT) ne nécessite généralement pas d'optimisation du rapport ARN/amorces et de sélection poly(A)+.Il est recommandé d'utiliser 0,5 μg d'oligo(dT) par système de réaction de 20 μl.oligo(dT)12-18 convient à la plupart des RT-PCR.Le système ThermoScript RT-PCR offre oligo(dT)20 en raison de sa meilleure stabilité thermique pour des températures d'incubation plus élevées.
Les amorces spécifiques au gène (GSP) sont les amorces les plus spécifiques pour l'étape de transcription inverse.Le GSP est un oligonucléotide antisens qui peut s'hybrider spécifiquement à la séquence cible de l'ARN, contrairement aux amorces aléatoires ou oligo(dT), qui s'hybrident à tous les ARN.Les mêmes règles utilisées pour concevoir des amorces PCR s'appliquent à la conception de GSP dans les réactions de transcription inverse.Le GSP peut être la même séquence que l'amorce d'amplification qui s'hybride à l'extrémité la plus en 3' de l'ARNm, ou le GSP peut être conçu pour s'hybrider en aval de l'amorce d'amplification inverse.Pour certains sujets amplifiés, plusieurs amorces antisens doivent être conçues pour une RT-PCR réussie car la structure secondaire de l'ARN cible peut empêcher la liaison de l'amorce.Il est recommandé d'utiliser 1 pmol de GSP antisens dans une réaction de synthèse du premier brin de 20 ul.
2. Augmentez la température d'incubation pour la transcription inverse:
Afin de tirer pleinement parti du plein avantage de la spécificité GSP, une transcriptase inverse avec une thermostabilité plus élevée doit être utilisée.Les transcriptases inverses thermostables peuvent être incubées à des températures plus élevées pour augmenter la stringence de la réaction.Par exemple, si un GSP s'annele à 55°C, la spécificité du GSP ne sera pas pleinement utilisée si l'AMV ou le M-MLV est utilisé pour la transcription inverse à une faible stringence de 37°C.Cependant, SuperScript II et ThermoScript peuvent être mis à réagir à 50°C ou plus, ce qui éliminera les produits non spécifiques générés à des températures plus basses.Pour une spécificité maximale, le mélange ARN/amorce peut être transféré directement de la température de dénaturation de 65 °C à la température d'incubation de la transcription inverse et ajouté à un mélange réactionnel 2 x préchauffé (démarrage à chaud de la synthèse d'ADNc).Cela aide à empêcher l'appariement de bases intermoléculaires à basse température.Les multiples transitions de température requises pour la RT-PCR peuvent être simplifiées en utilisant un thermocycleur.
3. Réduit la contamination de l'ADN génomique:
Une difficulté potentielle rencontrée avec la RT-PCR est la contamination de l'ADN génomique dans l'ARN.L'utilisation d'une bonne méthode d'isolement de l'ARN, telle que le réactif Trizol, réduira la quantité d'ADN génomique contaminant la préparation d'ARN.Pour éviter les produits dérivés de l'ADN génomique, l'ARN peut être traité avec de la DNase I de qualité amplification pour éliminer l'ADN contaminant avant la transcription inverse.La digestion à la DNase I a été terminée en incubant les échantillons dans 2,0 mM d'EDTA pendant 10 minutes à 65°C.L'EDTA peut chélater les ions magnésium, empêchant l'hydrolyse de l'ARN dépendant des ions magnésium à des températures élevées.
Afin de séparer l'ADNc amplifié des produits d'amplification d'ADN génomique contaminants, des amorces peuvent être conçues qui s'hybrident chacune pour séparer les exons.Les produits de PCR dérivés de l'ADNc seront plus courts que ceux dérivés de l'ADN génomique contaminé.De plus, une expérience de contrôle sans transcription inverse a été réalisée sur chaque matrice d'ARN pour déterminer si un fragment donné était dérivé d'ADN génomique ou d'ADNc.Le produit de PCR obtenu sans transcription inverse est issu du génome.
Heure de publication : 16 mai 2023








