Aperçu
Identification rapide des plantes transgéniques
Texte/Tong Yucheng
Opération expérimentale/Han Ying
Editeur/Wen Youjun
Mots/1600+
Temps de lecture suggéré/8-10 minutes
Identification rapide des plantes transgéniques
En tant que nouveau venu dans le laboratoire, il n'est pas judicieux de filtrer les plantes positives d'un groupe de plantes à faible taux de conversion.Premièrement, l'ADN doit être extrait d'un grand nombre d'échantillons un par un, puis les gènes étrangers seront détectés par PCR.Cependant, les résultats sont souvent des blancs et des bandes avec quelques items occasionnellement, mais il est impossible de déterminer s'il y a des détections manquées ou de fausses détections..Est-il très impuissant face à un tel processus expérimental et à de tels résultats ?Ne vous inquiétez pas, Brother vous apprend à filtrer facilement et avec précision les plantes transgéniques positives.
Étape 1: Concevoir des amorces de détection
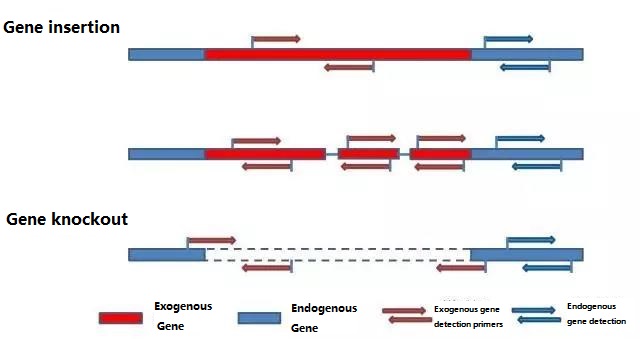
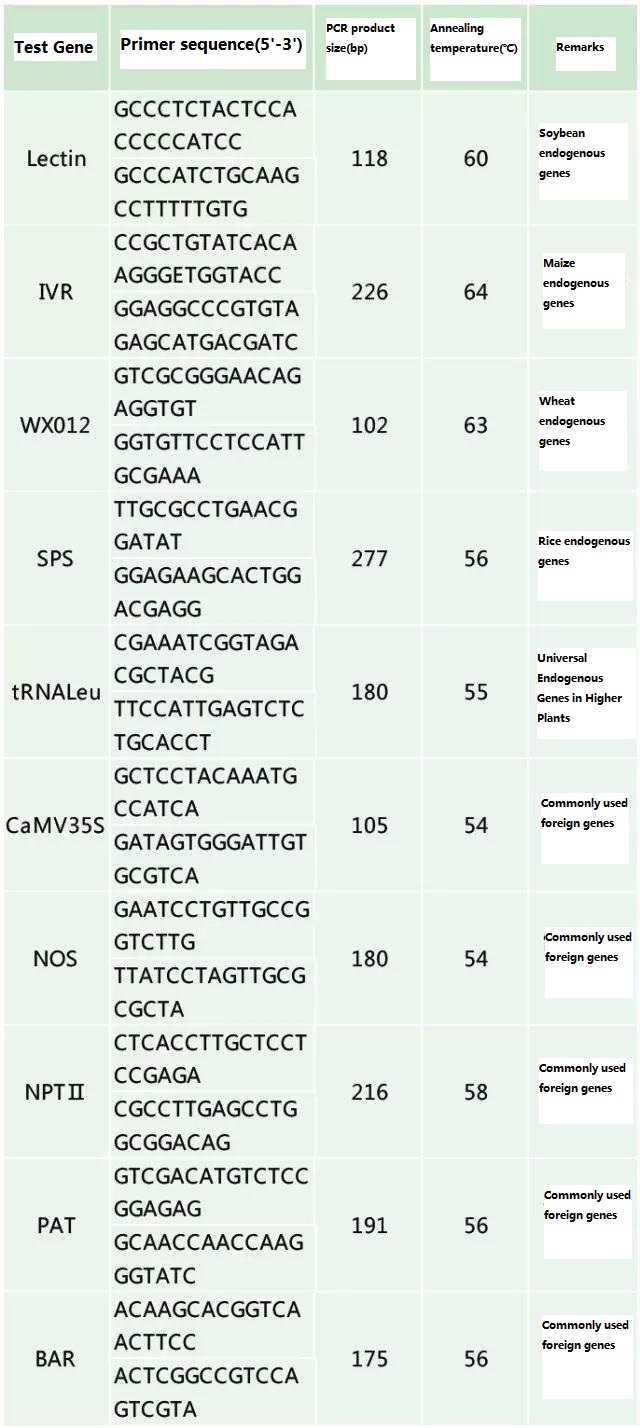
Déterminez le gène endogène et le gène exogène à détecter en fonction de l'échantillon à tester et sélectionnez une séquence représentative de 100 à 500 pb dans le gène pour la conception de l'amorce.De bonnes amorces peuvent garantir la précision des résultats de détection et raccourcir le temps de détection (voir l'annexe pour les amorces de détection couramment utilisées).
Note:
Les amorces nouvellement conçues doivent optimiser les conditions de réaction et vérifier l'exactitude, la précision et la limite de détection de la détection avant d'effectuer une détection à grande échelle.
Étape 2:Développer un protocole expérimental

Contrôle positif : Utilisez l'ADN purifié contenant le fragment cible comme matrice pour déterminer si le système et les conditions de réaction PCR sont normaux.
Contrôle négatif/blanc : Utiliser une matrice d'ADN ou ddH2O qui ne contient pas le fragment cible comme matrice pour détecter s'il existe une source de contamination dans le système PCR.
Contrôle de référence interne : utiliser la combinaison amorce/sonde du gène endogène de l'échantillon à tester pour évaluer si la matrice peut être détectée par PCR.
Note:
Des contrôles positifs, négatifs/blancs et des contrôles internes doivent être définis pour chaque test afin d'évaluer la validité des résultats expérimentaux.
Étape 3: Préparation de l'expérience

Avant utilisation, observez si la solution est mélangée uniformément.Si des précipitations sont détectées, elles doivent être dissoutes et mélangées conformément aux instructions avant utilisation.Le mélange 2 × PCR doit être pipeté et mélangé à plusieurs reprises avec une micropipette avant utilisation pour éviter une distribution inégale des ions.
Note:
Sortez les instructions et lisez-les attentivement, et préparez-vous avant l'expérience en stricte conformité avec les instructions.
Étape 4 : Préparer le système de réaction PCR
Selon le protocole expérimental, mélanger les amorces, H2O, mélangez 2 × PCR, centrifugez et distribuez-les dans chaque tube de réaction.
Note:
Pour les tests à grande échelle ou à long terme, il est recommandé d'utiliser un système de réaction PCR contenant l'enzyme UNG, qui peut efficacement éviter la contamination par aérosol causée par les produits PCR.
Étape 5 : Ajouter un modèle de réaction

Grâce à la technologie Direct PCR, aucun processus fastidieux de purification des acides nucléiques n'est nécessaire.La matrice d'échantillon peut être préparée en 10 minutes et ajoutée au système de réaction PCR correspondant.
Note:
La méthode de lyse a un meilleur effet de détection et le produit obtenu peut être utilisé pour de multiples réactions de détection.
5.1 : PCR directe des feuilles
Selon la taille de l'image dans le manuel, coupez le tissu foliaire d'un diamètre de 2-3 mm et placez-le dans le système de réaction PCR.
Remarque : Assurez-vous que les fragments de feuilles sont complètement immergés dans la solution de réaction PCR et n'ajoutez pas de tissu foliaire excessif.
5.2 : Méthode de lyse des feuilles
Coupez le tissu foliaire d'un diamètre de 5 à 7 mm et placez-le dans un tube à centrifuger.Si vous choisissez des feuilles matures, veuillez éviter d'utiliser les tissus de la nervure principale de la feuille.Pipeter 50ul de lysat de tampon P1 dans un tube à centrifuger pour s'assurer que le lysat peut immerger complètement le tissu foliaire, le placer dans un thermocycleur ou un bain de métal et lyser à 95°C pendant 5 à 10 minutes.


Ajouter 50ul de solution de neutralisation tampon P2 et bien mélanger.Le lysat résultant peut être utilisé comme matrice et ajouté au système de réaction PCR.
Remarque : La quantité de modèle doit être comprise entre 5 et 10 % du système PCR et ne doit pas dépasser 20 % (par exemple, dans un système PCR de 20 μl, ajouter 1 à 2 μl de tampon de lyse, pas plus de 4 μl).
Étape 6 : Réaction PCR

Après avoir centrifugé le tube de réaction PCR, placez-les dans un instrument PCR pour l'amplification.
Note:
La réaction utilise une matrice non purifiée pour l'amplification, de sorte que le nombre de cycles d'amplification est de 5 à 10 cycles de plus que lors de l'utilisation d'une matrice d'ADN purifiée.
Étape 7 : Détection de l'électrophorèse et analyse des résultats

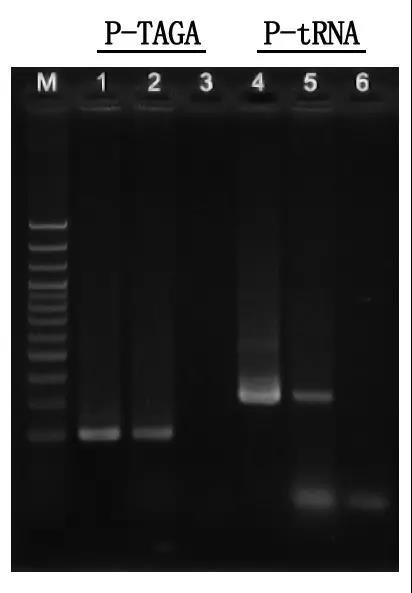
M : échelle d'ADN de 100 pb
1\4 : Méthode de l'ADN purifié
2\5 : Méthode de PCR directe
3\6 : contrôle vide
Contrôle de qualité:
Les résultats des tests des différents contrôles définis dans l'expérience doivent répondre aux conditions suivantes.Sinon, la cause du problème doit être analysée et le test doit être effectué à nouveau une fois le problème résolu.
Tableau 1. Résultats des tests normaux de divers groupes témoins

*Lorsque le plasmide est utilisé comme contrôle positif, le résultat du test du gène endogène peut être négatif
Jugement du résultat :
A. Le résultat du test du gène endogène de l'échantillon est négatif, ce qui indique que l'ADN adapté à la détection par PCR ordinaire ne peut pas être extrait de l'échantillon ou que l'ADN extrait contient des inhibiteurs de la réaction PCR et que l'ADN doit être extrait à nouveau.
B. Le résultat du test du gène endogène de l'échantillon est positif et le résultat du test du gène exogène est négatif, ce qui indique que l'ADN adapté à la détection par PCR ordinaire est extrait de l'échantillon et qu'il peut être jugé que le gène XXX n'est pas détecté dans l'échantillon.
C. Le résultat du test du gène endogène de l'échantillon est positif et le résultat du test du gène exogène est positif, indiquant que l'ADN adapté à la détection par PCR ordinaire a été extrait de l'échantillon et que l'ADN de l'échantillon contient le gène XXX.Des expériences de confirmation peuvent être réalisées ultérieurement.
Étape 8 : Concevoir des amorces de détection

Après l'expérience, utilisez une solution d'hypochlorite de sodium à 2 % et une solution d'éthanol à 70 % pour essuyer la zone expérimentale afin d'éviter la pollution de l'environnement.
annexe
Tableau 2. Amorces couramment utilisées pour la détection générale par PCR des plantes génétiquement modifiées
Document de référence:
SN/T 1202-2010, Méthode de détection qualitative par PCR des ingrédients végétaux génétiquement modifiés dans les aliments.
Annonce du ministère de l'Agriculture 1485-5-2010, Test des ingrédients de plantes génétiquement modifiées et de leurs produits - riz M12 et ses dérivés.
Heure de publication : 09 juin 2021








