La PCR (amplification en chaîne par polymérase) est l'une des technologies d'amplification in vitro de l'ADN, avec une histoire de plus de 30 ans.
La technologie PCR a été lancée par Kary Mullis de Cetus, États-Unis en 1983. Mullis a déposé une demande de brevet PCR en 1985 et a publié le premier article académique PCR sur la science la même année.Mullis a reçu le prix Nobel de chimie en 1993 pour ses travaux.
Principes de base de la PCR
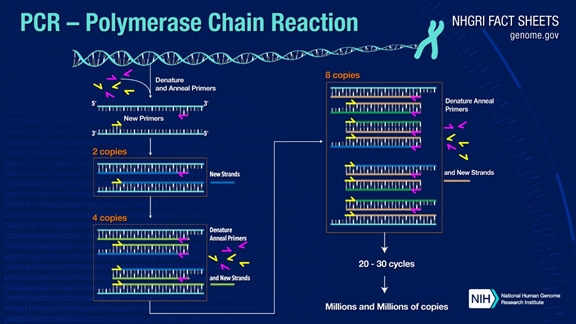
La PCR peut amplifier des fragments d'ADN cibles de plus d'un million de fois.Le principe est sous la catalyse de l'ADN polymérase, en utilisant l'ADN du brin parent comme matrice et une amorce spécifique comme point de départ pour l'extension.Il est répliqué in vitro par des étapes telles que la dénaturation, l'hybridation et l'extension.Le processus d'ADN du brin fille complémentaire de l'ADN matrice du brin parent.
Le processus standard de PCR est divisé en trois étapes :
1. Dénaturation : utilisez une température élevée pour séparer les doubles brins d'ADN.La liaison hydrogène entre les doubles brins d'ADN est rompue à haute température (93-98℃).
2.Annealing : Une fois l'ADN double brin séparé, abaissez la température afin que l'amorce puisse se lier à l'ADN simple brin.
3.Extension : L'ADN polymérase commence à synthétiser des brins complémentaires le long des brins d'ADN à partir des amorces liées lorsque la température est abaissée.Lorsque l'extension est terminée, un cycle est terminé et le nombre de fragments d'ADN double
En effectuant ces trois étapes en va-et-vient 25 à 35 fois, le nombre de fragments d'ADN augmentera de façon exponentielle.
L'ingéniosité de la PCR réside dans le fait que différentes amorces peuvent être conçues pour différents gènes cibles, de sorte que les fragments de gènes cibles peuvent être amplifiés en peu de temps.
Jusqu'à présent, la PCR peut être divisée en trois catégories, à savoir la PCR ordinaire, la PCR quantitative fluorescente et la PCR numérique.
La première génération de PCR ordinaire
Utilisez un instrument d'amplification PCR ordinaire pour amplifier le gène cible, puis utilisez une électrophorèse sur gel d'agarose pour détecter le produit, seule une analyse qualitative peut être effectuée.
Les principaux inconvénients de la PCR de première génération :
1.Sujet à une amplification non spécifique et à des résultats faussement positifs.
2.La détection prend beaucoup de temps et l'opération est lourde.
3. Seul un test qualitatif peut être effectué
PCR en temps réel de deuxième génération
La PCR en temps réel, également connue sous le nom de qPCR, utilise des sondes fluorescentes qui peuvent indiquer la progression du système de réaction, et surveille l'accumulation de produits amplifiés par l'accumulation de signaux fluorescents, et juge les résultats par la courbe de fluorescence.Il peut être quantifié à l'aide de la valeur Cq et de la courbe standard.
Parce que la technologie qPCR est réalisée dans un système fermé, la probabilité de contamination est réduite et le signal de fluorescence peut être surveillé pour une détection quantitative, il est donc le plus largement utilisé en pratique clinique et est devenu la technologie dominante en PCR.
Les substances fluorescentes utilisées dans la PCR quantitative fluorescente en temps réel peuvent être divisées en : sonde fluorescente TaqMan, balises moléculaires et colorant fluorescent.
1)Sonde fluorescente TaqMan :
Lors de l'amplification PCR, une sonde fluorescente spécifique est ajoutée tout en ajoutant une paire d'amorces.La sonde est un oligonucléotide et les deux extrémités sont marquées avec un groupe fluorescent reporter et un groupe fluorescent extincteur.
Lorsque la sonde est intacte, le signal fluorescent émis par le groupe rapporteur est absorbé par le groupe quenching ;lors de l'amplification par PCR, l'activité exonucléase 5′-3′ de l'enzyme Taq clive et dégrade la sonde, rendant le groupe rapporteur fluorescent et l'extincteur. Le groupe fluorescent est séparé, de sorte que le système de surveillance de la fluorescence puisse recevoir le signal de fluorescence, c'est-à-dire qu'à chaque fois qu'un brin d'ADN est amplifié, une molécule fluorescente se forme et l'accumulation du signal de fluorescence est complètement synchronisée avec la formation du produit PCR.
2) Colorant fluorescent SYBR :
Dans le système de réaction PCR, un excès de colorant fluorescent SYBR est ajouté.Une fois que le colorant fluorescent SYBR est incorporé de manière non spécifique dans le double brin d'ADN, il émet un signal fluorescent.La molécule de colorant SYBR qui n'est pas incorporée dans la chaîne n'émettra aucun signal fluorescent, assurant ainsi le signal fluorescent. L'augmentation des produits de PCR est complètement synchronisée avec l'augmentation des produits de PCR.Le SYBR ne se lie qu'à l'ADN double brin, de sorte que la courbe de fusion peut être utilisée pour déterminer si la réaction PCR est spécifique.
3) Balise moléculaire :
Il s'agit d'une sonde oligonucléotidique à double marquage tige-boucle qui forme une structure en épingle à cheveux d'environ 8 bases aux extrémités 5 et 3.Les séquences d'acide nucléique aux deux extrémités sont appariées de manière complémentaire, ce qui fait que le groupe fluorescent et le groupe d'extinction sont serrés.Fermer, aucune fluorescence ne sera produite.
Une fois le produit de PCR généré, pendant le processus d'hybridation, la partie médiane de la balise moléculaire est associée à une séquence d'ADN spécifique et le gène fluorescent est séparé du gène quencher pour produire une fluorescence.
Les principaux inconvénients de la PCR de seconde génération :
La sensibilité fait toujours défaut et la détection des spécimens à faible nombre de copies est imprécise.
Il y a l'influence de la valeur de fond et le résultat est sensible aux interférences.
Lorsqu'il y a des inhibiteurs de PCR dans le système de réaction, les résultats de détection sont sensibles aux interférences.
PCR numérique de troisième génération
La PCR numérique (DigitalPCR, dPCR, Dig-PCR) calcule le nombre de copies de la séquence cible grâce à la détection du point final et peut effectuer une détection quantitative absolue précise sans utiliser de contrôles internes ni de courbes standard.
La PCR numérique utilise la détection du point final et ne dépend pas de la valeur Ct (seuil de cycle), de sorte que la réaction PCR numérique est moins affectée par l'efficacité de l'amplification et la tolérance aux inhibiteurs de la réaction PCR est améliorée, avec une précision et une reproductibilité élevées.
En raison des caractéristiques de haute sensibilité et de haute précision, il n'est pas facilement interféré par les inhibiteurs de réaction PCR, et il peut réaliser une véritable quantification absolue sans produits standard, qui est devenu un point chaud de recherche et d'application.
Selon les différentes formes de l'unité de réaction, celle-ci peut être divisée en trois types principaux : les systèmes microfluidiques, puces et gouttelettes.
1) PCR numérique microfluidique, mdPCR :
Sur la base de la technologie microfluidique, la matrice d'ADN est séparée.La technologie microfluidique peut réaliser la nano-mise à niveau de l'échantillon ou la génération de gouttelettes plus petites, mais les gouttelettes nécessitent une méthode d'adsorption spéciale, puis combinées avec le système de réaction PCR.mdPCR a progressivement été adopté par d'autres méthodes de remplacement.
2) PCR numérique basée sur les gouttelettes, ddPCR :
Utilisez la technologie de génération de gouttelettes d'eau dans l'huile pour transformer l'échantillon en gouttelettes et divisez le système de réaction contenant des molécules d'acide nucléique en milliers de gouttelettes à l'échelle nanométrique, dont chacune ne contient pas la molécule cible d'acide nucléique à détecter, ou Contient une à plusieurs molécules cibles d'acide nucléique à tester.
3) PCR numérique sur puce, cdPCR :
Utilisez la technologie de voie de fluide intégrée pour graver de nombreux microtubes et microcavités sur des plaquettes de silicium ou de verre de quartz, et contrôler le débit de la solution à travers différentes vannes de contrôle, et diviser le liquide de l'échantillon en nanomètres de la même taille dans les puits de réaction pour la réaction PCR numérique pour obtenir une quantification absolue.
Les principaux inconvénients de la PCR de troisième génération :
Le matériel et les réactifs sont chers.
Les exigences de qualité des modèles sont élevées.Si la quantité de modèle dépasse la quantité de microsystème, il sera impossible de quantifier, et si elle est trop petite, la précision de quantification sera réduite.
Des faux positifs peuvent également être générés en cas d'amplification non spécifique.
Heure de publication : 30 juillet 2021