PCR, plusieurs PCR, PCR in situ, PCR inverse, RT-PCR, qPCR(1)–PCR
Nous allons trier les concepts, les étapes et les détails de divers PCR
Ⅰ. PCR
La réaction en chaîne par polymérase, appelée PCR, est une technologie de biologie moléculaire utilisée pour agrandir des fragments d'ADN spécifiques.Il peut être considéré comme une réplication spéciale de l'ADN in vitro.L'ADN polymérase (ADN polymérase I) a été découverte dès 1955, et le fragment de Klenow d'E. Coli, qui a une valeur expérimentale et pratique, a été découvert par le Dr H. Klenow au début des années 1970, mais parce que cette enzyme ne tolère pas la température, une température élevée peut la dégénérer, de sorte qu'elle ne rencontre pas la réaction en chaîne par polymérase avec une dégénérescence à haute température.Les enzymes utilisées aujourd'hui (appelées Taq polymérase), ont été isolées de Thermus aquaticus, une bactérie thermale en 1976. Sa caractéristique est qu'elle peut résister à des températures élevées et est une enzyme idéale, mais elle est largement utilisée après les années 1980.Le concept original du prototype primitif original de PCR est similaire à la réparation et à la copie de gènes, qui a été proposé par le Dr KJell Kleppe en 1971. Il a publié la première copie de gène simple et à court terme (similaire aux deux premières réactions de cycle de PCR).Le PCR développé aujourd'hui a été développé par le Dr Kary B. Mullis en 1983. Le Dr Mullis a servi des entreprises de PE cette année-là, de sorte que le PE a un statut spécial dans l'industrie de la PCR.Le Dr Mullis a officiellement publié le premier article connexe avec Saiki et d'autres en 1985. Depuis lors, l'utilisation de la PCR se fait à des milliers de kilomètres par jour, et on peut dire que la qualité des articles connexes rend de nombreuses autres méthodes de recherche désagréables.Par la suite, la technologie PCR est largement utilisée dans la recherche scientifique biologique et les applications cliniques, devenant la technologie la plus importante de la recherche en biologie moléculaire.Mullis a également remporté le prix Nobel de chimie en 1993.
PCRPrincipe
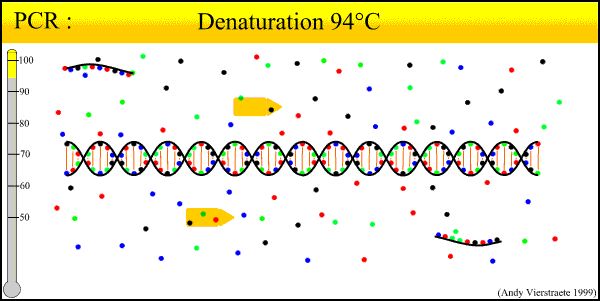
Le principe de base de la technologie PCR est similaire au processus de réplication naturelle de l'ADN, et sa spécificité dépend de l'amorce oligonucléotidique qui est complémentaire aux deux extrémités de la séquence cible.La PCR est composée de trois étapes de réactions de base de dégénérescence-annelage-extension : ①Dégénérescence de l'ADN matrice : après que l'ADN matrice est chauffé à environ 93 °C pendant un certain temps, la solution d'ADN double pour l'ADN à double chaîne formée par l'amplification PCR de l'ADN matrice en partant, en fait une chaîne unique afin qu'elle puisse être combinée avec l'amorce pour préparer la réaction suivante.②L'hybridation (composé) de l'ADN matrice et de l'amorce : une fois que l'ADN matrice est chauffé et dégénéré en une seule chaîne, la température chute à environ 55 °C.La séquence complémentaire de l'amorce et de la matrice ADN monocaténaire.③L'extension de l'amorce : la matrice d'ADN - la liaison de l'amorce est basée sur l'action de la TaqDNA polymérase, avec le dNTP comme matière première de réaction.Conservez le principe de réplication, synthétisez une nouvelle chaîne de copie semi-réservée qui complète la chaîne d'ADN modèle, et répétez le cycle de dégénérescence-recuit-extension trois processus peuvent obtenir plus de «chaîne de copie semi-réservée», et cette nouvelle chaîne est à nouveau disponible Devenez un modèle pour le prochain cycle.Il faut 2 à 4 minutes pour boucler la boucle, le gène cible peut être amplifié plusieurs millions de fois en 2 à 3 heures.
StandardPCRSystème de réaction
| Taq ADN polymérase | 2,5 μl |
| Mg2+ | 1,5 mmol/L |
| Tampon d'amplification 10× | 10μl |
| 4 mélanges de dNTP | 200μl |
| ADN modèle | 0,1~2μg |
| Apprêt | 10~100μl |
| Ajouter de l'eau à double ou triple vapeur | 100 μl |
Cinq éléments de la réaction PCR
Il existe principalement cinq types de substances impliquées dans la réaction PCR, à savoir l'amorce, l'enzyme, le dNTP, la matrice et le tampon (Mg2+ est nécessaire).[Procédure PCR]
Le processus standard de PCR est divisé en trois étapes
1. Dégénérescence de l'ADN (90°C-96°C) : matrices d'ADN à double chaîne sous l'action thermique, rupture des liaisons hydrogène, formant un ADN à chaîne unique.
2. Recuit (25℃ -65℃): La température du système est réduite, l'amorce est combinée avec la matrice d'ADN pour former une double chaîne locale.
3. Extension (70 ℃ -75 ℃): Sous l'action de l'enzyme Taq (environ 72 ° C, la meilleure activité), le dNTP est utilisé comme matière première, s'étend de l'extrémité 5 'de l'amorce → extrémité 3 ', la synthèse et la matrice se complètent mutuellement chaîne d'ADN.
Chaque cycle est dénaturé, annelé et prolongé, doublant la teneur en ADN.À l'heure actuelle, en raison de la courte zone d'amplification, certaines PCR peuvent être répliquées en très peu de temps même si l'activité enzymatique Taq n'est pas optimale, elle peut donc être modifiée en deux étapes, c'est-à-dire que l'annelage et l'extension peuvent être effectués à 60°C-65°C en même temps.Afin de réduire le processus de levage et de refroidissement et d'améliorer la vitesse de réponse.
Caractéristiques de la réaction PCR
● Haute spécificité
Les facteurs décisifs spécifiques de la réponse PCR sont : ①La combinaison spécifique de l'amorce et de l'ADN matrice.②Le principe de l'appariement de base.③La fidélité de la réaction de synthèse de la TaqDNA polymérase.④La spécificité et la prudence du gène cible.
La bonne combinaison d'amorces et de modèles est la clé.La liaison de l'amorce et de la matrice et l'extension de la chaîne de l'amorce sont basées sur le principe de l'appariement des bases alcalines.La fidélité des réactions de synthèse de polymérase et la résistance à haute température de l'ADN polymérase Taq pour réaliser la liaison (composé) de la matrice et de l'amorce dans la réaction peuvent être réalisées à une température plus élevée.La spécificité de la combinaison est fortement augmentée.Le clip peut maintenir un degré élevé d'exactitude.En sélectionnant une région génétique cible avec une conservativité élevée et une conservativité élevée, sa spécificité est plus élevée.
● Haute sensibilité
Le volume de production de produits PCR est augmenté par index, ce qui peut étendre le modèle de départ de Picker (PG = 10-12) pour augmenter le niveau de microcontrôleur au niveau des microgrammes (μg = -6).Une cellule cible peut être détectée à partir de 1 million de cellules ;dans la détection des virus, la sensibilité de la PCR peut atteindre 3 RFU (unités formées de points vides) ;le taux de détection minimum en bactériologie est de 3 bactéries.
● Simple et rapide
La réflexion PCR utilise une ADN polymérase Taq à haute température, qui ajoute la solution de réaction en une seule fois, c'est-à-dire une réaction de dégénérescence-recuit-extension sur la solution d'amplification d'ADN et le pot de bain-marie.Généralement, la réaction d'amplification est terminée en 2 à 4 heures.Les produits augmentés sont généralement analysés par épée électrique, et n'ont pas à utiliser d'isotopes, pas de pollution radioactive, et une promotion facile.
● La pureté de l'échantillon est faible
Il n'est pas nécessaire de séparer les virus ou les bactéries et les cellules de culture.Les produits bruts d'ADN et l'ARN peuvent être utilisés comme amplificateurs.La détection par amplification de l'ADN peut être utilisée directement à l'aide d'échantillons cliniques tels que le sang, les liquides corporels, le liquide de lavage de la toux, les cheveux, les cellules et les tissus vivants.
PCRproblèmes communs
● Faux négatif, pas de bandes amplifiées
Les étapes clés de la réaction PCR comprennent : ① la préparation des acides nucléiques matrices, ② la qualité et la spécificité des amorces, ③ la qualité des enzymes ④ les conditions du cycle PCR.Trouver la raison doit également être analysé et étudié pour les liens ci-dessus.
Modèles : ① Le modèle contient diverses protéines, ② Le modèle contient un inhibiteur de l'enzyme Taq, ③ La protéine dans le modèle n'est pas éliminée, en particulier la protéine de groupe dans le chromosome.⑤ La dégénérescence des acides nucléiques du démineur n'est pas complète.Lorsque la qualité des enzymes et des amorces est bonne, il n'y a pas de bande d'amplification, ce qui est très probablement le traitement digestif des échantillons.Il y a quelque chose qui ne va pas avec le processus d'extraction de l'acide nucléique matrice, donc pour préparer une solution de digestion efficace et stable, sa procédure doit être fixée et non modifiée arbitrairement.
Inactivation enzymatique : une nouvelle enzyme ou des enzymes anciennes et nouvelles doivent être utilisées ensemble pour analyser si l'activité enzymatique est perdue ou insuffisante, ce qui entraîne des faux négatifs.A noter que l'enzyme Taq ou le bromure d'éthidium est parfois oublié.
Amorce : la qualité de l'amorce, la concentration de l'amorce et si la concentration des deux amorces est symétrique.C'est une raison courante de l'échec de la PCR ou la bande croissante n'est pas idéale et a tendance à se diffuser.Il y a des problèmes avec la qualité des amorces de certains numéros de lot.Les deux amorces ont une concentration élevée et une concentration faible, provoquant une amplification asymétrique à faible efficacité.Les contre-mesures sont : ① Sélectionnez une bonne amorce pour synthétiser les unités.② La concentration de l'amorce dépend non seulement de la valeur OD, mais prête également attention au liquide d'origine de l'amorce pour faire une électrophorèse sur gel de sucre d'agar.Il doit y avoir une zone de bande d'amorce et la luminosité des deux amorces doit être généralement cohérente.Belt, la PCR peut échouer à ce moment, et il doit être résolu avec l'unité de synthèse d'amorces.Si un apprêt est élevé, la luminosité est faible et sa concentration doit être équilibrée lorsqu'il est dilué.③ L'apprêt doit être payé et stocké à une concentration élevée pour éviter le gel multiple ou les pièces de réfrigération à long terme du réfrigérateur, ce qui entraînera la détérioration et la dégradation de l'apprêt.④ La conception de l'amorce est déraisonnable, comme la longueur de l'amorce est insuffisante, et le di cluster est formé entre les amorces.
Concentration Mg2+ : La concentration en ions Mg2+ a un impact important sur l'efficacité de l'amplification PCR.Une concentration excessive peut réduire le sexe opposé de l'amplification PCR.Si la concentration est trop faible, la sortie d'amplification PCR entraînera même l'échec de l'amplification PCR sans la bande d'expansion.
Changement de volume de réaction : le volume utilisé dans l'amplification PCR est de 20 ul, 30 ul et 50 ul ou 100 uL, le grand volume de l'application pour l'amplification PCR est défini en fonction des différents objectifs de la recherche scientifique et des tests cliniques.Après avoir réalisé de petits volumes comme 20ul, il est nécessaire de faire une condition de cordon lors de la réalisation de la taille, sinon cela échouera.
Raisons physiques : La transformation est très importante pour l'amplification par PCR.Si la température de dégénérescence est basse, le temps de dégénérescence est court, il est susceptible de se produire en faux négatifs ;une température de recuit trop basse peut provoquer une amplification non spécifique et réduire l'efficacité de l'amplification spécifique.Affecte fortement la combinaison d'amorces et de matrices pour réduire l'efficacité de l'amplification par PCR.Parfois, il est nécessaire d'utiliser des thermomètres standard pour détecter la variabilité, le recuit et la température prolongée dans l'extension ou le cuiseur hydrosoluble, qui est l'une des raisons de l'échec de la PCR.
Variantes de séquence cible : Si la séquence cible se produit, une mutation ou une délétion, la combinaison du prototype et de la matrice est combinée, ou en raison de l'absence d'une séquence cible, l'amorce et la matrice perdront la séquence complémentaire, et son amplification par PCR ne réussira pas.
● Faux positif
La bande d'amplification PCR semble cohérente avec la bande de séquence cible, et parfois sa bande est plus nette et plus élevée.
La conception de l'amorce n'est pas appropriée : la séquence d'amplification sélectionnée et la séquence d'amplification sans but ont des homologues, donc lors de l'amplification par PCR, les produits de PCR amplifiés sont des séquences sans but.La séquence cible est trop courte ou l'amorce est trop courte, et elle est sujette aux faux positifs.Besoin d'être repensé.
Pollution croisée de la séquence cible ou des produits d'amplification : Il y a deux raisons à cette pollution : Premièrement, la pollution croisée de l'ensemble du génome ou de grands segments, conduisant à des faux positifs.Ce type de faux positif peut être résolu par les méthodes suivantes : Soyez prudent et doux pendant le fonctionnement pour éviter que la séquence cible ne soit inhalée dans le pistolet à échantillon ou ne s'éclabousse du tube centrifuge.À l'exception des enzymes et des substances qui ne supportent pas les températures élevées, tous les réactifs ou équipements doivent être désinfectés à haute pression.Les tuyaux centrifuges et les échantillons doivent être utilisés en même temps.Si nécessaire, avant d'ajouter des échantillons, le tube de réaction et le réactif sont exposés aux rayons ultraviolets pour détruire l'acide nucléique existant.Deuxièmement, de petits fragments dans la pollution de l'air.Ces petits fragments sont plus courts que la séquence cible, mais ils présentent une certaine homologie.Il peut être épissé les uns avec les autres.Après avoir complété les amorces, le produit PCR peut être étendu, ce qui entraînera une production de faux positifs.Il peut être utilisé pour réduire ou éliminer la méthode Nest PCR.
● Apparaître une bande d'amplification non spécifique
Les bandes apparues après amplification PCR sont incohérentes avec la taille attendue, ou grandes ou petites, ou à la fois, ou en même temps, des bandes d'amplification spécifiques et des bandes d'amplification non spécifiques.L'émergence de bandes non spécifiques est la suivante : Premièrement, les amorces sont complémentaires incomplètes de la séquence cible, ou la polymérisation de l'amorce pour former un cluster di.La seconde est que la concentration en ions MG2+ est trop élevée, la température de recuit est trop basse et le nombre de cycles de PCR est lié.Deuxièmement, la qualité et la quantité d'enzymes.Souvent, les enzymes de certaines sources sont sujettes à des bandes non spéciales et les enzymes de l'autre source ne se produisent pas.Parfois, une amplification non spécifique des enzymes se produit également.Les contre-mesures sont les suivantes : reconcevoir les attractifs si nécessaire.Réduisez la quantité d'enzyme ou remplacez l'enzyme d'une autre source.Réduisez la quantité de primaire, augmentez la quantité de modèles de manière appropriée et réduisez le nombre de cycles.Augmentez correctement la température de recuit ou utilisez la méthode à deux points de température (dégénérescence à 93 °C, recuit et extension à environ 65 °C).
● Apparaître un remorquage floconneux ou un ruban adhésif
L'amplification par PCR semble parfois être appliquée ou décortiquée ou tapis-comme une ceinture.Pour cette raison, en raison de la quantité excessive d'enzymes ou de la mauvaise qualité de l'enzyme, la concentration de dNTP est trop élevée, la concentration de Mg2+ est trop élevée, la température de recuit est trop basse et le nombre de cycles est trop élevé.Les contre-mesures sont : ①Réduire la quantité d'enzymes ou changer l'enzyme d'une autre source.②Réduire la concentration de dNTP ③Réduire correctement la concentration de Mg2+.④Augmentez la quantité de modèles et réduisez le nombre de cycles.
Produits connexes
◮ Fidélité supérieure : 6 fois celle de l'enzyme Taq ordinaire ;
◮ Vitesse d'amplification plus rapide
◮ Plus d'adaptabilité des modèles
◮ Efficacité d'amplification supérieure
◮ La tolérance environnementale est plus forte : placé à 37°C pendant une semaine, maintien de plus de 90% d'activité ;
◮ Il possède une activité ADN polymérase 5'→3' et une activité exonucléase 5'→3', sans activité exonucléase 3'→5'.
Le système de réaction unique et l'ADN polymérase Taq à haute efficacité confèrent à la réaction PCR une efficacité, une spécificité et une sensibilité d'amplification supérieures.
RT-qPCR Easyᵀᴹ (une étape)-SYBR Green I
◮ Le kit en une étape permet la transcription inverse et la qPCR deux réactions dans le même tube, il suffit d'ajouter de l'ARN modèle, des amorces PCR spécifiques et du ddH sans RNase2O.
◮ Le kit peut rapidement et efficacement analyser quantitativement l'ARN viral ou tracer l'ARN.
◮ Le kit utilise un réactif de transcription inverse Foregene unique et l'ADN polymérase Foregene HotStar Taq combinés à un système de réaction unique pour améliorer efficacement l'efficacité de l'amplification et la spécificité de la réaction.
◮ Le système de réaction optimisé donne à la réaction une sensibilité de détection plus élevée, une stabilité thermique plus forte et une meilleure tolérance.
◮ RT-qPCR facileTM(One Step) - Le kit SYBR Green I est livré avec un colorant de référence interne ROX, qui peut être utilisé pour éliminer le bruit de fond du signal et les erreurs de signal entre les puits, ce qui est pratique pour les clients à utiliser dans différents modèles d'instruments PCR quantitatifs.
RT FacileTMII (Master Premix pour synthèse d'ADNc premier brin pourPcr en temps réel)
-Capacité efficace d'éliminer l'ADNg, qui peut éliminer l'ADNg dans le modèle en 2 minutes.
-Système de transcription inverse efficace, il ne faut que 15 minutes pour terminer la synthèse du premier brin d'ADNc.
-Modèles complexes : les modèles à haute teneur en GC et à structure secondaire complexe peuvent également être inversés avec une grande efficacité.
-Système de transcription inverse à haute sensibilité, les modèles de niveau pg peuvent également obtenir un ADNc de haute qualité.
-Le système de transcription inverse a une stabilité thermique élevée, la température de réaction optimale est de 42℃ et il a toujours de bonnes performances de transcription inverse à 50℃.
Heure de publication : 18 mars 2023