Émergence du SRAS-CoV-2 B.1.1.7 Lignée—
États-Unis, 29 décembre 2020–12 janvier 2021
Summer E. Galloway, PhD 1 ;Prabasaj Paul, PhD 1 ;Duncan R. MacCannell, PhD 2 ;Michael A. Johansson, Ph. D. 1 ;
John T. Brooks, MD 1 ;Adam MacNeil, Ph. D. 1 ;Rachel B. Slayton, PhD 1 ;Suxiang Tong, PhD 1 ;Benjamin J. Silk, PhD 1 ;Gregory L. Armstrong, MD 2 ;
Matthew Biggerstaff, ScD 1 ;Vivien G. Dugan, PhD
Le 15 janvier 2021, ce rapport a été publié en tant que MMWRPublication anticipée sur le site Web du MMWR (https://www.cdc.gov/mmwr).
Le 14 décembre 2020, le Royaume-Uni a signaléune variante préoccupante (VOC) du SARS-CoV-2, lignée B.1.1.7,également appelé VOC 202012/01 ou 20I/501Y.V1.* LeOn estime que la variante B.1.1.7 est apparue en septembre2020 et est rapidement devenu le dominant en circulationVariante du SRAS-CoV-2 en Angleterre (1).B.1.1.7 a étédétecté dans plus de 30 pays, dont les États-Unis.Commedu 13 janvier 2021, environ 76 cas de B.1.1.7 ontété détecté dans 12 États américains.Plusieurs sources de preuvesindiquent que B.1.1.7 est transmis plus efficacement queautres variantes du SARS-CoV-2 (1–3).La trajectoire modélisée decette variante aux États-Unis affiche une croissance rapide au début de 2021,devenant la variante prédominante en mars.AugmentéLa transmission du SRAS-CoV-2 pourrait menacer les soins de santé tendusressources, nécessitent une mise en œuvre étendue et plus rigoureusedes stratégies de santé publique (4), et augmenter le pourcentage deimmunité de la population requise pour le contrôle de la pandémie.Priseles mesures visant à réduire la transmission maintenant peuvent réduire le potentielimpact de B.1.1.7 et laisser un temps critique pour augmenter la vaccinationcouverture.Collectivement, une surveillance génomique amélioréecombiné avec le respect continu des règles publiques efficacesmesures sanitaires, dont la vaccination, la distanciation physique,l'utilisation de masques, l'hygiène des mains, l'isolement et la quarantaine,être essentiel pour limiter la propagation du SRAS-CoV-2, le virusqui cause la maladie à coronavirus 2019 (COVID-19).Stratégiquedépistage des personnes sans symptômes mais à risque plus élevé deinfection, comme ceux exposés au SRAS-CoV-2 ou qui ontcontacts fréquents et inévitables avec le public, fournit un autrepossibilité de limiter la propagation en cours.
Surveillance génomique mondiale et partage open-source rapideing de séquences du génome viral ont facilité le temps quasi réeldétection, comparaison et suivi de l'évolution du SRAS-CoV-2variantes qui peuvent éclairer les efforts de santé publique pour contrôler lapandémie.Alors que certaines mutations du génome viralémergent puis reculent, d'autres pourraient conférer un avantage sélectiftage au variant, y compris une transmissibilité améliorée, de sorte queune telle variante peut rapidement dominer d'autres variantes en circulation.
Au début de la pandémie, des variantes du SRAS-CoV-2 contenantla mutation D614G dans la protéine de pointe (S) qui augmentel'avidité de liaison aux récepteurs est rapidement devenue dominante chez de nombreuxrégions géographiques (5,6).À la fin de l'automne 2020, plusieurs pays ont signalé avoir détectéVariantes du SRAS-CoV-2 qui se propagent plus efficacement.En outreà la variante B.1.1.7, les variantes notables incluent le B.1.351lignée détectée pour la première fois en Afrique du Sud et la lignée récemment identifiéeB.1.1.28 sous-clade (renommée"P.1”) détecté chez quatre voyageursdu Brésil lors d'un dépistage de routine au Haneda (Tokyo)aéroport.§ Ces variantes portent une constellation de muta génétiquetions, y compris dans le domaine de liaison au récepteur de la protéine S,qui est essentiel pour se lier à l'angiotensine de la cellule hôteconvertir le récepteur de l'enzyme-2 (ACE-2) pour faciliter le virusentrée.Les preuves suggèrent que d'autres mutations trouvées dans cesvariantes pourraient conférer non seulement une transmissibilité accrue, maispeut également affecter les performances de certains diagnostics en temps réeltranscription inversée–réaction en chaîne par polymérase (RT-PCR)dosages¶ et réduire la sensibilité aux anticorps neutralisants(2,3,5–dix).Un rapport de cas récent a documenté le premier cas deRéinfection du SARS-CoV-2 au Brésil avec une variante du SARS-CoV-2qui contenait la mutation E484K,** qui a été démontréepour réduire la neutralisation par les sérums de convalescence et les monoclonauxanticorps (9,10).
Ce rapport se concentre sur l'émergence de la variante B.1.1.7aux Etats-Unis.Au 12 janvier 2021, ni leB.1.351 ni les variantes P.1 n'ont été détectées dans leÉtats-Unis.Pour plus d'informations sur le SRAS-CoV-2 émergentvariantes préoccupantes, CDC maintient une page Web dédiée àfournir des informations sur les variantes émergentes du SRAS-CoV-2.††
B.1.1.7 lignée (20I/501Y.V1)
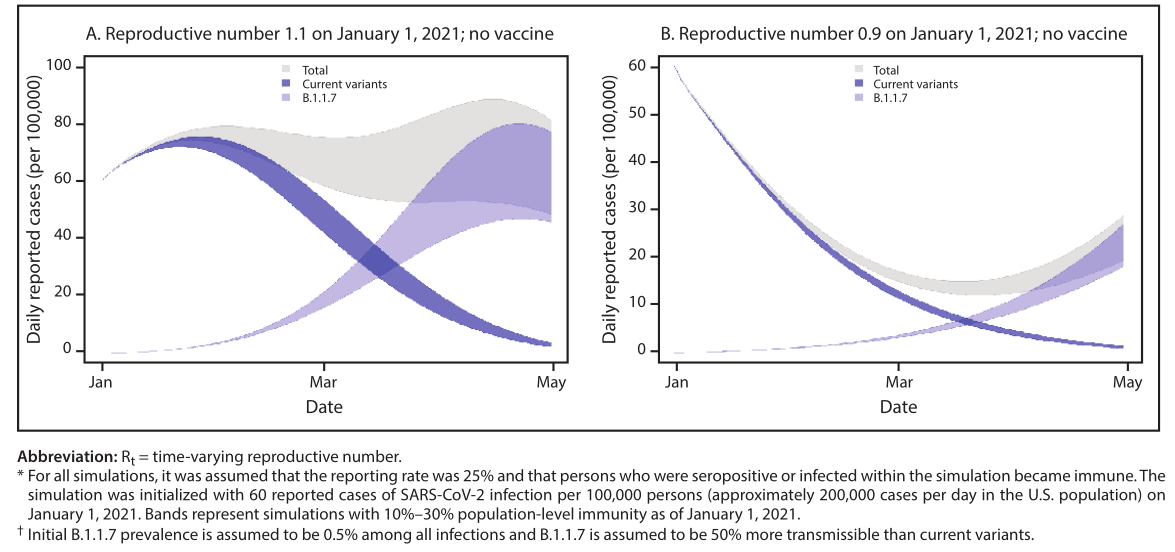
Le variant B.1.1.7 porte une mutation dans la protéine S(N501Y) qui affecte la conformation de la liaison aux récepteursdomaine.Cette variante a 13 autres mutations définissant la lignée B.1.1.7 (tableau), dont plusieurs sont dans la protéine S,dont une suppression aux positions 69 et 70 (del69–70) queévolué spontanément dans d'autres variantes du SRAS-CoV-2 et estsupposé augmenter la transmissibilité (2,7).La suppressionaux positions 69 et 70 provoque un échec de la cible du gène S (SGTF)dans au moins une RT-PCR–test de diagnostic basé surTest ThermoFisher Taq Path COVID-19, le B.1.1.7 varifourmi et autres variantes avec le del69–70 produisent un négatifrésultat pour la cible du gène S et un résultat positif pour les deux autrescibles);SGTF a servi de mandataire au Royaume-Unipour identifier les cas B.1.1.7 (1).Plusieurs sources de données indiquent que B.1.1.7 est plusefficacement transmis par rapport aux autres SARS-CoV-2variantes circulant au Royaume-Uni.régions du Royaume-Uni avecune proportion plus élevée de séquences B.1.1.7 avait une épidémie plus rapidecroissance que d'autres domaines, les diagnostics avec SGTF ont augmentéplus rapide que les diagnostics non SGTF dans les mêmes zones, et unune proportion plus élevée de contacts ont été infectés par des patients indexavec des infections B.1.1.7 que par les patients index infectés avecautres variantes (1,3).La variante B.1.1.7 a le potentiel d'augmenter le pan américaintrajectoire démique dans les mois à venir.Pour illustrer cet effet,un modèle compartimenté simple à deux variantes a été développé.La prévalence actuelle aux États-Unis de B.1.1.7 parmi tous lesvirus est inconnu, mais on pense qu'il est inférieur à 0,5 % sur la base de lanombre limité de cas détectés et données SGTF (8).Pourmodèle, les hypothèses initiales comprenaient une prévalence B.1.1.7de 0,5 % parmi toutes les infections, l'immunité au SRAS-CoV-2 deinfection antérieure de 10 %–30%, un reproducteur variable dans le tempsnombre (R t ) de 1,1 (transmission atténuée mais croissante)ou 0,9 (diminution de la transmission) pour les variantes actuelles, et une incidence signalée de 60 cas pour 100 000 personnes par jour le1er janvier 2021. Ces hypothèses ne représentent pas précisémentn'importe quel endroit aux États-Unis, mais indiquent plutôt une généralisation deconditions communes à travers le pays.Le changement de R t surtemps résultant de l'immunité acquise et de l'augmentation de la prévalencelence de B.1.1.7, a été modélisée, avec le B.1.1.7 R t supposéêtre une constante 1,5 fois le R t des variantes actuelles, sur la base deestimations initiales du Royaume-Uni (1,3).Ensuite, l'impact potentiel de la vaccination a été modéliséen supposant que 1 million de doses de vaccin ont été administrées parjour commençant le 1er janvier 2021, et que 95% d'immunitéa été atteint 14 jours après réception de 2 doses.Spécifiquement,l'immunité contre l'infection par les variants actuels ou leLa variante B.1.1.7 a été supposée, bien que l'efficacité etla durée de la protection contre l'infection reste incertaine,parce que ceux-ci n'étaient pas le critère d'évaluation principal des essais cliniquespour les premiers vaccins.Dans ce modèle, la prévalence de B.1.1.7 est initialement faible, mais parce queil est plus transmissible que les variantes actuelles, il présentecroissance rapide début 2021, devenant la variété prédominantefourmi en mars (Figure 1).Que ce soit la transmission du courantvariantes augmente (R initial t = 1,1) ou diminue lentement(R t initial = 0,9) en janvier, B.1.1.7 entraîne un changement substantieldans la trajectoire de transmission et une nouvelle phase de croissance exponentiellecroissance.Avec la vaccination qui protège contre l'infection, leles trajectoires épidémiques précoces ne changent pas et B.1.1.7 se propagentse produit encore (Figure 2).Cependant, après que B.1.1.7 devient levariante dominante, sa transmission a été considérablement réduite.L'effet de la vaccination sur la réduction de la transmission dans le procheterme était le plus élevé dans le scénario dans lequel la transmission étaitdécroît déjà (R t initial = 0,9) (Figure 2).Les premiers efforts quipeut limiter la propagation de la variante B.1.1.7, telle que universelle etrespect accru des stratégies d'atténuation de la santé publique,laissera plus de temps à la vaccination en cours pour atteindre des niveaux plus élevésimmunité au niveau de la population.
Discussion
Actuellement, il n'y a pas de différence connue dans les résultats cliniquesassocié aux variants SARS-CoV-2 décrits ;cependant,un taux de transmission plus élevé conduira à plus de cas, augmentantle nombre total de personnes qui ont besoin de soins cliniques, exacersoulager le fardeau d'un système de santé déjà mis à rude épreuve,et entraînant plus de décès.Surveillance génomique continuepour identifier les cas B.1.1.7, ainsi que l'émergence d'autresvariantes préoccupantes aux États-Unis, est important pour laRéponse de santé publique COVID-19.Alors que les résultats de la SGTFpeut aider à identifier les cas B.1.1.7 potentiels qui peuvent être confirméspar séquençage, identifiant les variants prioritaires qui ne présentent pasLa SGTF s'appuie exclusivement sur la surveillance basée sur les séquences.
|
Désignation de la variante | Première identification | Mutations caractéristiques (protéine : mutation) | Nombre de cas actuels à séquence confirmée | Nombre de pays avec séquences | ||
| Emplacement | Date | États-Unis | Mondial | |||
| B.1.1.7 (20I/501Y.V1) | Royaume-Uni | septembre 2020 | ORF1ab : T1001I, A1708D, I2230T, del3675–3677 SGF S : del69–70 HV, del144 Y, N501Y, A570D, D614G, P681H, T761I, S982A, D1118H ORF8 : Q27stop, R52I, Y73C N : D3L, S235F | 76 | 15 369 | 36 |
| B.1.351 (20H/501Y.V2) | Afrique du Sud | octobre 2020 | ORF1ab : K1655N E : P71L N : T205I S:K417N, E484K, N501Y, D614G, A701V | 0 | 415 | 13
|
| P.1 (20J/501Y.V3 | Brésil et Japon | janv. 2021 | ORF1ab : F681L, I760T, S1188L, K1795Q, del3675–3677 SGF, E5662D S : L18F, T20N, P26S, D138Y, R190S, K417T, E484K, N501Y, D614G, H655Y, T1027I ORF3a : C174G ORF8 : E92K ORF9 : Q77E ORF14 : V49L N : P80R | 0 | 35 | 2
|
Abréviations : del = suppression ;E = protéine d'enveloppe ;N = protéine de nucléocapside ;ORF = cadre de lecture ouvert ;S = protéine de pointe.
L'expérience au Royaume-Uni et les modèles B.1.1.7présentés dans ce rapport illustrent l'impact d'unevariable peut avoir sur le nombre de cas dans une population.Lel'augmentation de la transmissibilité de ce variant nécessite unemise en œuvre rigoureuse et combinée de la vaccination et de la mitigamesures de protection (p. ex., distanciation, port du masque et hygiène des mains)pour contrôler la propagation du SRAS-CoV-2.Ces mesures serontplus efficaces s'ils sont institués plus tôt que tardpour ralentir la propagation initiale de la variante B.1.1.7.Des efforts pourpréparer le système de santé à de nouvelles poussées de cas sontgaranti.Une transmissibilité accrue signifie également qu'une plus grandeque la couverture vaccinale prévue doit être atteinte pouratteindre le même niveau de contrôle des maladies pour protéger le publicpar rapport aux variantes moins transmissibles.En collaboration avec des universitaires, industriels, étatiques, territoriaux,partenaires tribaux et locaux, CDC et autres agences fédéralescoordonnent et améliorent la surveillance génomique etefforts de caractérisation des virus à travers les États-Unis.CDCcoordonne les efforts de séquençage des États-Unis via le SARS-CoV-2Séquençage pour la réponse aux urgences de santé publique,Épidémiologie et surveillance (SPHERES)§§consortium,qui comprend environ 170 institutions participantes et promeut le partage ouvert de données pour faciliter l'utilisation du SRAS-CoV-2données de séquence.Pour suivre l'évolution virale du SRAS-CoV-2, le CDC estmettre en œuvre une surveillance génomique à multiples facettes pour comprendreles processus épidémiologiques, immunologiques et évolutifsqui façonnent les phylogénies virales (phylodynamique);guider l'éclosionenquêtes;et faciliter la détection et la caractérisationtion des réinfections possibles, des cas de percée vaccinale etvariantes virales émergentes.En novembre 2020, CDC a établile programme national de surveillance des souches SARS-CoV-2 (NS3)pour améliorer la représentativité du SARS-CoV-2 domestiqueséquences.Le programme collabore avec 64 publics américainsdes laboratoires de santé pour soutenir un système de surveillance génomique ;NS3 constitue également une collection de spécimens de SARS-CoV-2 and séquences pour soutenir la réponse de santé publique et scientifiquerecherche pour évaluer l’impact de mutations préoccupantes surcontre-mesures médicales recommandées existantes.CDC aa également conclu des contrats avec plusieurs grands laboratoires cliniques commerciauxconservateurs pour séquencer rapidement des dizaines de milliers de SARS-CoV-2–spécimens positifs chaque mois et a financé sept études universitairesinstitutions pour mener une surveillance génomique en partenariatavec les agences de santé publique, ajoutant ainsi considérablement àla disponibilité de données de surveillance génomique en temps opportun provenant deles États Unis.Outre ces initiatives nationales,de nombreuses agences de santé publique nationales et locales séquencent
FIGURE 1. Trajectoires d'incidence des cas simulées* des variantes actuelles du SRAS-CoV-2 et de la variante B.1.1.7,†en supposant qu'il n'y a pas de vaccination communautaireet soit R t initial = 1,1 (A) soit R t initial = 0,9 (B) pour les variantes actuelles—États-Unis, janvier–avril 2021
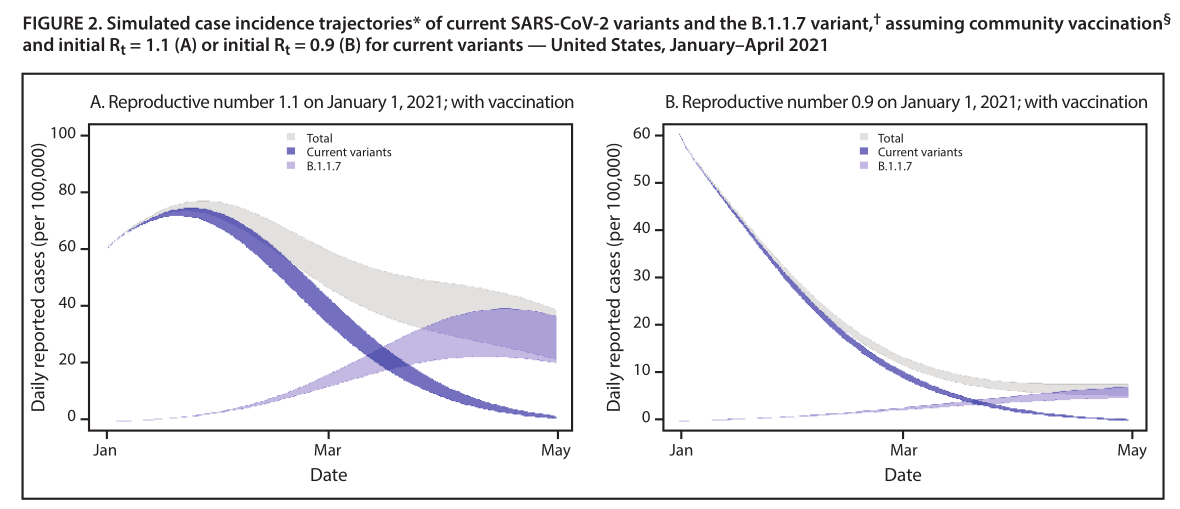


SARS-CoV-2 pour mieux comprendre l'épidémiologie locale etsoutenir la réponse de santé publique à la pandémie.Les conclusions de ce rapport sont soumises à au moins trois limitestations.Tout d'abord, l'ampleur de l'augmentation de transmissibilaux États-Unis par rapport à celle observée aux États-UnisLe Royaume-Uni reste incertain.Deuxièmement, la prévalence deB.1.1.7 aux États-Unis est également inconnu pour le moment, maisla détection des variants et l'estimation de la prévalence améliorerontavec les efforts de surveillance renforcés des États-Unis.Enfin, la mitiga localeles mesures de performance sont également très variables, ce qui entraîne des variations dansRt.Les résultats spécifiques présentés ici sont basés sur des simulationset supposé aucun changement dans les mesures d'atténuation au-delà du 1er janvier.La transmissibilité accrue de la guerre des variantes B.1.1.7dénonce la mise en œuvre rigoureuse de stratégies de santé publique pourréduire la transmission et atténuer l'impact potentiel de B.1.1.7,acheter du temps critique pour augmenter la couverture vaccinale.CDCles données de modélisation montrent que l'utilisation universelle et une conformité accrueance avec des mesures d'atténuation et la vaccination sont cruciales pourréduire considérablement le nombre de nouveaux cas et de décès dans lemois à venir.De plus, le test stratégique des personnes sanssymptômes de la COVID-19, mais qui présentent un risque accru deinfection par le SRAS-CoV-2, offre une autre occasion delimiter la propagation continue.Collectivement, une surveillance génomique amélioréelance combinée à une conformité accrue avec la santé publiquestratégies d'atténuation, y compris la vaccination, la distanciation physiquel'utilisation de masques, l'hygiène des mains, l'isolement et la quarantaine,seront essentiels pour limiter la propagation du SRAS-CoV-2 etprotéger la santé publique.
Remerciements
Membres du Sequencing for Public Health EmergencyConsortium d'intervention, d'épidémiologie et de surveillance ;étatique et localelaboratoires de santé publique;Association des laboratoires de santé publique ;Équipe d'intervention du CDC COVID-19 ;Direction des virus respiratoires,Division des maladies virales, CDC. Formulaire du Comité des éditeurs de revues médicales pour la divulgation d'un potentielles conflits d'intérêts.Aucun conflit d'intérêts potentiel n'a été divulgué.
Les références
1. Santé publique Angleterre.Investigation of novel SARS-CoV-2 variant: variant of concern 202012/01, briefing technique 3. Londres, Royaume-Uni : Public Health England ;2020. https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_data/file/950823/Variant_of_Concern_VOC_202012_01_Technical_Briefing_3_-_England.pdf
2. Kemp SA, Harvey WT, Datir RP et al.Émergence et transmission récurrentes d'une délétion de pointe SARS-CoV-2 ΔH69/V70.bioRxiv[Préimpression mise en ligne le 14 janvier 2021].https://www.biorxiv.org/content/10.1101/2020.12.14.422555v4
3. Volz E, Mishra S, Chand M, et al.Transmission de la lignée B.1.1.7 du SRAS-CoV-2 en Angleterre : aperçu de la liaison des données épidémiologiques et génétiques.medRxiv [Preprint mis en ligne le 4 janvier 2021].https://www.medrxiv.org/content/10.1101/2020.12.30.20249034v2
4. Honein MA, Christie A, Rose DA, et al. ;Équipe d'intervention du CDC COVID-19.Résumé des orientations pour les stratégies de santé publique visant à faire face aux niveaux élevés de transmission communautaire du SRAS-CoV-2 et des décès associés, décembre 2020. MMWR Morb Mortal Wkly Rep 2020;69:1860–7.PMID:33301434 https://doi.org/10.15585/mmwr.mm6949e2
5. Volz E, Hill V, McCrone JT, et al. ;Consortium COG-UK.Évaluer les effets de la mutation D614G du pic SARS-CoV-2 sur la transmissibilité et la pathogénicité.Cellule 2021;184:64–75.e11.PMID : 33275900 https://doi.org/10.1016/j.cell.2020.11.020
6. Korber B, Fischer WM, Gnanakaran S, et al. ;Groupe de génomique Sheffield COVID-19.Suivi des changements dans le pic de SRAS-CoV-2 : preuve que le D614G augmente l'infectiosité du virus COVID-19.Cellule
2020;182:812–27.PMID : 32697968 https://doi.org/10.1016/j.cell.2020.06.043
7. McCarthy KR, Rennick LJ, Namnulli S, et al.Les délétions naturelles dans la glycoprotéine de pointe du SRAS-CoV-2 échappent aux anticorps.bioRxiv [Preprint mis en ligne le 19 novembre 2020].https://www.biorxiv.org/content/
10.1101/2020.11.19.389916v18.Washington NL, White S, Schiabor KM, Cirulli ET, Bolze A, les schémas d'abandon du gène Lu JT.S dans les tests SARS-CoV-2 suggèrent la propagation de la mutation H69del/V70del aux États-Unis.medRxiv [Preprint mis en ligne le 30 décembre 2020].https://www.medrxiv.org/content/10.1101/2020.12.24.20248814v1
9. Weisblum Y, Schmidt F, Zhang F, et al.Échapper aux anticorps neutralisants par les variantes de la protéine de pointe du SRAS-CoV-2.eLife 2020;9:e61312.PMID:33112236 https://doi.org/10.7554/eLife.61312
10. Greaney AJ, Loes AN, Crawford KHD, et al.Cartographie complète des mutations du domaine de liaison au récepteur SARS-CoV-2 qui affectent la reconnaissance par les anticorps sériques humains polyclonaux.bioRxiv [Preprint mis en ligne le 4 janvier 2021].https://www.biorxiv.org/content/10.1101/2020.12.31.425021v1
Heure de publication : 11 février 2021








