Le vaccin ARNm de Pfizer contre le COVID a ravivé la passion pour l'utilisation de l'acide ribonucléique (ARN) comme cible thérapeutique.Cependant, cibler l'ARN avec de petites molécules est extrêmement difficile.
L'ARN n'a que quatre éléments constitutifs : l'adénine (A), la cytosine (C), la guanine (G) et l'uracile (U) qui remplace la thymine (T) présente dans l'ADN.Cela fait de la sélectivité des médicaments un obstacle presque insurmontable.En revanche, il existe 22 acides aminés naturels qui composent les protéines, ce qui explique pourquoi la plupart des médicaments ciblant les protéines ont une sélectivité relativement bonne.
Structure et fonction de l'ARN
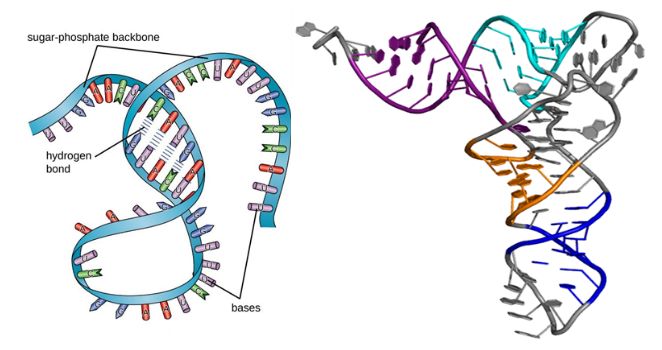
Comme les protéines, les molécules d'ARN ont des structures secondaires et tertiaires, comme le montre la figure ci-dessous.Bien qu'il s'agisse de macromolécules à chaîne unique, leur structure secondaire prend forme lorsque l'appariement de bases provoque des renflements, des boucles et des hélices.Ensuite, le repliement tridimensionnel conduit à la structure tertiaire de l'ARN, essentielle à sa stabilité et à sa fonction.
Figure 1. Structure de l'ARN
Il existe trois types d'ARN :
- ARN messager (ARNm)transcrit l'information génétique de l'ADN et est transféré sous forme de séquence de bases sur le ribosome ;je
- ARN ribosomique (ARNr)fait partie des organites synthétisant les protéines appelées ribosomes, qui sont exportées vers le cytoplasme et aident à traduire les informations de l'ARNm en protéines ;
- ARN de transfert (ARNt)est le lien entre l'ARNm et la chaîne d'acides aminés qui compose la protéine.
Cibler l'ARN comme cible thérapeutique est très attractif.Il a été constaté que seulement 1,5 % de notre génome est finalement traduit en protéine, tandis que 70 % à 90 % est transcrit en ARN.Les molécules d'ARN sont les plus importantes pour tous les organismes vivants.Selon le « dogme central » de Francis Crick, le rôle le plus critique de l'ARN est de traduire l'information génétique de l'ADN en protéines.En outre, les molécules d'ARN ont également d'autres fonctions, notamment :
- Agissant comme molécules adaptatrices dans la synthèse des protéines;je
- Servir de messager entre l'ADN et le ribosome;je
- Ils sont porteurs d'informations génétiques dans toutes les cellules vivantes ;je
- Favoriser la sélection ribosomale des acides aminés corrects, nécessaires à la synthèse de nouvelles protéinesin vivo.
Antibiotiques
Bien qu'il ait été découvert dès les années 1940, le mécanisme d'action de nombreux antibiotiques n'a été élucidé qu'à la fin des années 1980.Il a été constaté qu'une grande partie des antibiotiques agissent en se liant aux ribosomes bactériens pour les empêcher de fabriquer les protéines appropriées, tuant ainsi les bactéries.
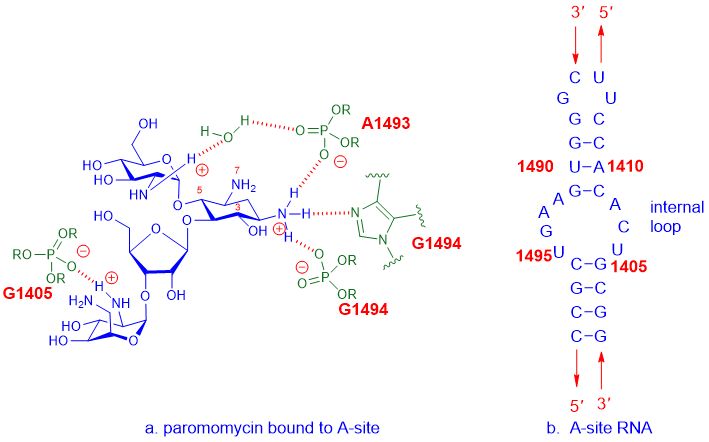
Par exemple, les antibiotiques aminoglycosides se lient au site A de l'ARNr 16S, qui fait partie de la sous-unité du ribosome 30S, puis interfèrent avec la synthèse des protéines pour interférer avec la croissance bactérienne, entraînant finalement la mort cellulaire.Le site A fait référence au site aminoacyle, également connu sous le nom de site accepteur d'ARNt.L'interaction détaillée entre les médicaments aminoglycosides, tels queparomomycine, et le site A deE. coliL'ARN est montré ci-dessous.
Figure 2. L'interaction entre la paromomycine et le site A deE. coliARN
Malheureusement, de nombreux inhibiteurs du site A, y compris les médicaments aminoglycosides, présentent des problèmes de sécurité tels que la néphrotoxicité, l'ototoxicité dose-dépendante et irréversible spécifique.Ces toxicités sont le résultat d'un manque de sélectivité des médicaments aminosides pour la reconnaissance des petites molécules d'ARN.
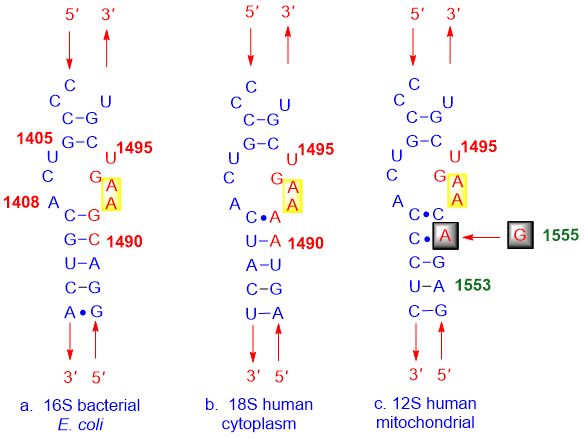
Comme le montre la figure ci-dessous : (a) la structure de la bactérie, (b) la membrane cellulaire humaine et (c) le site A mitochondrial humain sont très similaires, ce qui permet aux inhibiteurs du site A de se lier à chacun d'eux.
Figure 3. La liaison non sélective de l'inhibiteur du site A
Les antibiotiques tétracyclines inhibent également le site A de l'ARNr.Ils inhibent sélectivement la synthèse des protéines bactériennes en se liant de manière réversible à une région hélicoïdale (H34) sur la sous-unité 30S complexée avec Mg2+.
D'autre part, les antibiotiques macrolides se lient près du site de sortie (site E) du tunnel ribosomique bactérien pour les peptides naissants (NPET) et le bloquent partiellement, inhibant ainsi la synthèse des protéines bactériennes.Enfin, les antibiotiques oxazolidinone tels quele linézolide(Zyvox) se lient à une fente profonde dans la sous-unité ribosomique bactérienne 50S, qui est entourée de nucléotides d'ARNr 23S.
Oligonucléotides antisens (ASO)
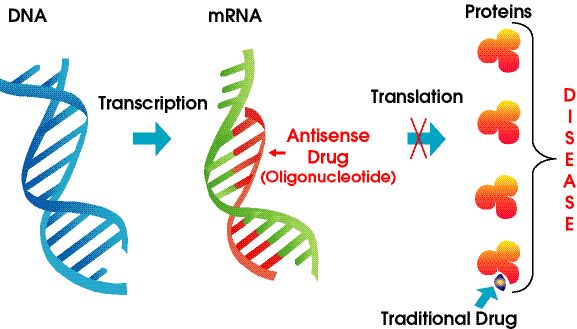
Les médicaments antisens sont des polymères d'acide nucléique chimiquement modifiés qui ciblent l'ARN.Ils s'appuient sur l'appariement de bases Watson-Crick pour se lier à l'ARNm cible, ce qui entraîne un silençage génique, un blocage stérique ou une altération de l'épissage.Les ASO peuvent interagir avec les pré-ARN dans le noyau cellulaire et les ARNm matures dans le cytoplasme.Ils peuvent cibler les exons, les introns et les régions non traduites (UTR).À ce jour, plus d'une douzaine de médicaments ASO ont été approuvés par la FDA.
Figure 4. Technologie antisens
Médicaments à petites molécules ciblant l'ARN
En 2015, Novartis a annoncé avoir découvert un régulateur d'épissage SMN2 appelé Branaplam, qui améliore l'association de l'U1-pré-ARNm et sauve les souris SMA.
En revanche, Risdiplam (Evrysdi) de PTC/Roche a été approuvé par la FDA en 2020 pour le traitement de la SMA.Comme Branaplam, Risdiplam agit également en régulant l'épissage des gènes SMN2 pertinents pour produire des protéines SMN fonctionnelles.
Dégradateurs d'ARN
RBM signifie protéine de motif de liaison à l'ARN.Essentiellement, l'indole sulfonamide est un adhésif moléculaire.Il recrute sélectivement RBM39 dans l'ubiquitine ligase CRL4-DCAF15 E3, favorisant la polyubiquitination de RBM39 et la dégradation des protéines.L'épuisement génétique ou la dégradation médiée par les sulfamides de RBM39 induit des anomalies d'épissage importantes à l'échelle du génome, conduisant finalement à la mort cellulaire.
Les ARN-PROTAC sont développés pour dégrader les protéines de liaison à l'ARN (RBP).PROTAC utilise un lieur pour connecter le ligand E3 ligase au ligand ARN, qui se lie à l'ARN et aux RBP.Étant donné que RBP contient des domaines structuraux qui peuvent se lier à des séquences oligonucléotidiques spécifiques, RNA-PROTAC utilise une séquence oligonucléotidique comme ligand pour la protéine d'intérêt (POI).Le résultat final est la dégradation des RBP.
Récemment, le professeur Matthew Disney de la Scripps Institution of Oceanography a inventé l'ARNchimères ciblant la ribonucléase (RiboTAC).RiboTAC est une molécule hétérofonctionnelle qui relie un ligand RNase L et un ligand ARN avec un lieur.Il peut recruter spécifiquement la RNase L endogène vers des cibles d'ARN spécifiques, puis éliminer avec succès l'ARN en utilisant le mécanisme de dégradation des acides nucléiques cellulaires (RNase L).
Au fur et à mesure que les chercheurs en apprendront davantage sur l'interaction entre les petites molécules et les cibles d'ARN, davantage de médicaments utilisant cette méthode émergeront à l'avenir.
Heure de publication : 02 août 2023